


 |
 |
 |
068/2012 Proyecto Certificación Genética del Origen de Materiales Reproductivos de Coigüe mediante Herramientas Moleculares y Modelos Ecológicos |
||
| Resumen y Objetivos | Investigadores | Marco Teórico y Métodos | Resultados del Proyecto |
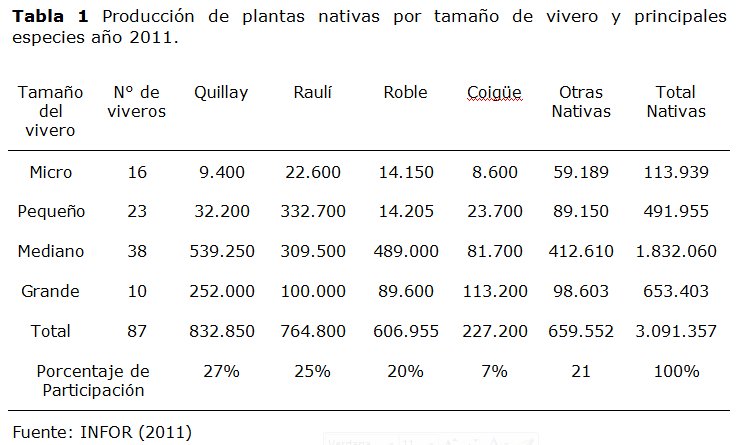
Marco Teórico del Proyecto Antecedentes Históricos La conservación, utilización y manejo de especies arbóreas nativa tiene una serie de beneficios ambientales, sociales y económicos. Su protección y uso sustentable favorece la conservación de los recursos genéticos para las futuras generaciones, junto al cuidado de los recursos hídricos y de suelo, contribuye notablemente a la atenuación del clima local, reduce el impacto de emisiones de gases invernadero y ayuda a la conservación de los hábitats naturales y de la diversidad biológica. Desde el punto de vista social tiene una serie de funciones recreativas y de valoración del paisaje. Adicionalmente, la posibilidad de generar una variada gama de productos madereros y no madereros a partir de especies forestales nativas como Nothofagus alpina (Raulí), Nothofagus obliqua (Roble), Nothofagus dombeyi (Coigüe) y Quillaja saponaria (Quillay) entre otras, ha incentivado notablemente su uso en las últimas décadas, convirtiéndolas en alternativas reales de diversificación forestal. El Instituto Forestal de Chile (INFOR) en su misión de apoyar a las instituciones públicas y agentes económicos privados del sector forestal, a través de la generación de información y tecnologías para una eficiente asignación y uso sostenible de los recursos forestales, ha establecido desde 1962 múltiples ensayos desde la I Región de Tarapacá a la XII Región de Magallanes, con más de 300 especies tanto exóticas como nativas. A partir del año 1997, INFOR inicia el programa de mejoramiento genético de especies nativas, con Nothofagus pumilio (Lenga), N. alpina (raulí), N. dombeyi, N. obliqua, Laurelia sempervirens (Laurel) y Eucryphia cordifolia (Ulmo), a través del cual hoy se están habilitando fuentes productoras de semillas genéticamente mejoradas. En 2008 se promulga la Ley de Recuperación del Bosque Nativo y Fomento Forestal, que busca fundamentalmente proteger, recuperar y mejorar las especies nativas del país, asegurando su sustentabilidad forestal, mediante planes de manejo y preservación. Se crea un Fondo de Conservación, Recuperación y Manejo Sustentable del Bosque Nativo que es administrado por el Ministerio de Agricultura. A través de él se otorgan bonificaciones para solventar, en parte, el costo de las actividades que favorezcan la regeneración, recuperación o protección de formaciones xerofíticas (adaptadas a la aridez) de alto valor ecológico o de bosques nativos de preservación. Antes de la entrada del nuevo milenio, Donoso et al. (1999) señalaron que dado el potencial de crecimiento y calidad, las especies más plantadas en el centro sur de Chile son Raulí y Coigüe. En menor medida se utilizaba Ulmo y Roble para fines madereros, y en zonas de recuperación de bosques Laurel, Olivillo y Lingue. En últimos 20-25 años se han plantado Nothofagus con una mayor tecnología silvícola, perfeccionando en primer lugar el primer eslabón de la cadena productiva que es la calidad de planta a utilizar, produciéndolas en viveros con altos estándares de producción. A partir del año 2000 se registran las primeras experiencias con establecimiento intensivo de plantaciones (manejo físico del suelo, fertilización y control de maleza), en dichas situaciones se han registrado crecimientos notables tanto en coihue como en Raulí, los cuales bordean en promedio 2 cm anuales en diámetro y 1,5 m en altura (Soto y Ríos, 2009). A pesar de estos bueno resultados, la demanda y con ello la producción de plantas forestales nativas sigue siendo marginal (2%) en relación a especies exóticas de rápido crecimiento como Pinus radiata (Pino radiata), Eucalyptus globulus y Eucalyptus nitens (95%). Dentro de las especies nativas, algunas presentan una mayor demanda de plantas (Tabla 1). Tal es el caso del Quillay, Raulí y Roble, especies que en conjunto hoy representan más del 70% de las plantas producidas (INFOR 2011), cambiado notablemente su importancia en cuanto a producción de plantas en relación al año 2002 cuando aun no aparecían entre las 3 especies más comercializadas (Castillo y Moreno 2002). |
||
 |
||
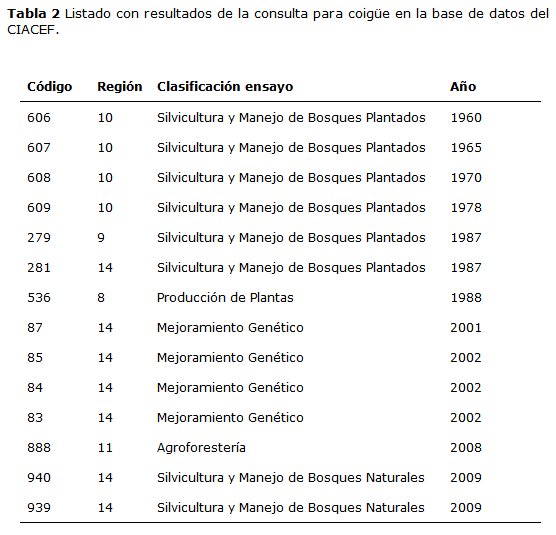
Según los antecedentes que se presentan a continuación, esta tendencia no sería casual, sino que puede estar directamente relacionada con la implementación de programas de mejoramiento avanzados. La totalidad de los esfuerzos de mejoramiento genético para especies forestales del bosque nativo de chileno hasta el año 2000 aparecen claramente detallados en Castillo y Moreno (2002), donde se puede notar que roble, raulí y quillay recibieron gran atención. Adicionalmente para las dos primeras especies, en el año 2000 se inicia un ambicioso e innovador proyecto financiado por Fondo Regional de Tecnología Agropecuaria (FONTAGRO) en el que participó el Instituto Nacional de Investigaciones Agrícolas (INIA), INFOR, Corporación Nacional Forestal (CONAF), Instituto Nacional de Tecnología Agropecuarias (INTA) de Argentina entre otras instituciones. El objetivo general fue evaluar la variabilidad genética de Roble y Raulí en Chile y Argentina mediante el uso de marcadores moleculares (RAPDs, cpADN) e isoenzimáticos para la fijación de criterios de conservación, mejoramiento genético, reforestación, manejo y aprovechamiento. El análisis conjunto de ambos tipos de marcadores permitió la identificación de áreas prioritarias para la conservación (según criterios genéticos) a partir de lo cual surgieron pedidos oficiales a las autoridades competentes en cada caso para; i) la re-categorización en el caso de áreas de jurisdicción de Parques Nacionales o ii) la declaración como área de reserva en el caso de bosques de propiedad privada. También se identificaron zonas de transferencia de semilla para el futuro manejo silvícola de la región. En el caso del Quillay la alta demanda de plantas puede estar explicada por su amplia distribución geográfica, multiplicidad de productos obtenidos a partir de sus extractos y un programa de mejoramiento genético dirigido por la Universidad Mayor a partir de 2004. El cual se basó en el desarrollo industrial de extractos de quillay y selección genotípica. Para cumplir el segundo objetivo, se realizó una evaluación de la diferenciación genecológica del Quillay en Chile, muestreando individuos a lo largo de toda la distribución de la especie, entre la IV y la IX Región. Esta evaluación permitió preseleccionar un grupo de individuos en función de su rendimiento industrial, los que fueron clonados y usados en ensayos de terreno. Paralelamente, en conjunto con el INIA se hizo un estudio de la diversidad genética que presenta esta colección representativa del germoplasma de quillay, utilizando la técnica de análisis genético denominada “Amplified Fragment Length Polymorphism” (AFLP). Estos estudios permitieron describir el desempeño de los genotipos preseleccionados, sentar bases respecto a la incidencia genética y ambiental en la producción de los extractos del Quillay, y además, seleccionar individuos para la implementación de un huerto semillero clonal. Diferente ha sido la situación de coigüe, especie para la cual no se encuentran en las bases de datos nacionales iniciativas como las llevadas a cabo para raulí, roble y quillay. Se señala esto luego de conversaciones con expertos de la Universidad de Concepción, INFOR y CONAF, información que fue ratificada luego de realizar una búsqueda en el Centro de Información de Experiencias de Adaptación y Crecimiento de Especies Forestales en Chile (CIACEF; www.infor.cl/ciacef/). El CIACEF es una base de datos orientada a poner en conocimiento y uso público la información de 48 años de investigación sobre especies forestales en Chile con fines productivos y de experimentación científica. La información entregada para Coigüe se resume en la Tabla 2. Se detectan cuatro ensayos relacionados con mejoramiento genético. Estos ensayos son parte de un proyecto FONDEF destinado a generar fuentes de semilla genéticamente mejorada (huertos semilleros clonales y áreas productoras de semillas). No obstante, Coigüe no ha logrado ponerse al nivel del Raulí, Roble o Quillay en cuanto a la producción y demanda de plantas.
Identificación del problema La menor producción de plantas de Coigüe podría estar explicada por un significativo déficit en la oferta de semillas de calidad que garanticen el traspaso del potencial productivo de la especie a los futuros proyectos de forestación o reforestación. En este sentido, el mercado de las semillas forestales en Chile (y en gran medida de las plantas), se caracteriza por un alto nivel de informalidad, compuesto por muchos proveedores pequeños que entregan productos de origen desconocido y muchas veces inadecuado para el lugar de plantación. Es posible que la falta de programas de mejoramiento avanzados en Coigüe unido a la necesidad urgente de normativas que permitan certificar la calidad genética de las semillas forestales en general, estén afectando de manera particular a esta especie. |
||
 |
||
El caso del Coigüe; alternativa de desarrollo forestal sustentable La capacidad de las especies latifoliadas para producción de madera, favorecer actividades asociadas a la conservación de suelos y agua, mejoramiento de hábitat para la vida silvestre y en general para el desarrollo sostenible del sector forestal productivo justifica su creciente uso en Chile (CONAF 2005, 2006; INFOR, 2011) como en otras zonas templadas (Barret y Jacobs 2006). Entre las latifoliadas nativas de Chile; Roble, Raulí y Coigüe están entre las más valoradas. Luego de un gran trabajo de recopilación de información, Donoso y Soto (2010) resumen los resultados al menos 30 años investigación en plantaciones con especies nativas en centro-sur de Chile. Se consideran distintos esquemas de establecimiento, condiciones ambientales y de manejo cultural. Sobre la base de dicha información se puede concluir que en general, en sitios más mésicos (depresión intermedia y en ambas precordilleras hasta los 300 m s.n.m.), Coigüe se desarrolla muy bien en diferentes ambientes, a diferencia de Raulí que requiere de condiciones específicas de luz, agua, nutrientes y interacciones con otras especies arbóreas y arbustivas. Dentro de estas experiencias, se pueden recopilar muy pocas plantaciones sometidas a una oportuna y adecuada silvicultura intermedia que demuestren el verdadero potencial de crecimiento y calidad de losproductos maderables a extraer. Usando control de la vegetación competidora y fertilización inicial en el establecimiento de plantaciones de Raulí y Coigüe permiten aventurar modelos silvícolas teóricos muy auspiciosos (2 cm en diámetro y 1,5 m en altura en sus primeros 5 años de desarrollo), pudiendoser comparables con Pino radiata en un índice de sitio 28 (IS: altura a una edad clave de 20 años), lo que disminuiría las rotaciones de 40-50 años del Coigüe a 25-35 años, dependiendo de la calidad de sitio. Respecto a la bonificación forestal, se afirma que la tabla de costo para plantaciones nativas de la Ley 28.283 de Fomento y Recuperación del Bosque Nativo, es insuficiente para pagar los costos de una plantación con alta tecnología silvícola a campo abierto. Sin embargo, cuando este punto mejore se van a demandar plantas de la mejor calidad genética para obtener los más altos rendimientos, ya que el negocio forestal es de largo plazo y concentra los mayores costos al inicio. El uso de plantas de calidad deficiente o generadas a partir de semillas de un origen inadecuado para el sitio de plantación tiene un impacto negativo en la rentabilidad de un proyecto de forestación o reforestación. Seguramente la marginalidad en las tasas de plantación de especies como el Coigüe se ven acentuadas por la ausencia de una oferta de semillas de calidad, lo que finalmente se traduce en pocos incentivos para su utilización, dado que resulta poco probable alcanzar el potencial productivo.
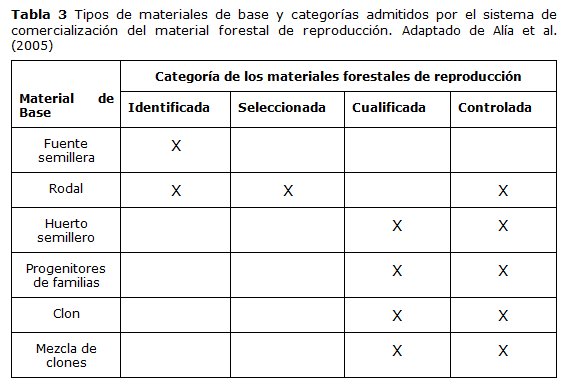
Normativa de comercialización de material forestal de reproducción La "calidad de la planta" involucra atributos morfológicos, fisiológicos y genéticos que determinan la supervivencia, adaptabilidad, crecimiento y productividad de un cultivo. Con excepción de algunos caracteres morfológicos, tales atributos no son perceptibles a simple vista, situación que genera una falla de transparencia de mercado y provoca incertidumbre en los compradores respecto al desempeño que exhibirán las plantas una vez establecida la plantación. Por consiguiente, la única garantía que le queda al usuario, en cuanto a la identidad que aparece en el documento de compra, es esperar que haya funcionado correctamente todo el flujo de controles establecidos en la cosecha de los frutos, el procesado y transformación en semillas, en el almacenamiento y comercialización y finalmente en la producción de planta. El mercado nacional de semillas nativas forestales se caracteriza por ser informal y por la comercialización de materiales de origen desconocido (INFOR 2011), lo que finalmente afecta la productividad y adaptabilidad de las especies. Dada la importancia de los materiales forestales de reproducción (frutos, semillas, plantas y partes de plantas utilizados en el proceso de regeneración natural o artificial) se han establecido normas nacionales e internacionales que regulan el proceso de comercialización, desde los tipos de materiales de basede los que se pueden obtener, a las características que deben reunir estos materiales. De esta forma se busca disminuir la incertidumbre y otorgar cierta garantía a los compradores respecto de la calidad del producto que adquieren. Estos mecanismos de certificación son comunes en diversos países. En Europa hay dos sistemas de comercialización; i) para los estados miembros de la Unión Europea (UE) existe una directiva sobre comercialización de materiales forestal de reproducción (Sistema UE) de aplicación obligatoria para todos ellos: ii) con carácter abierto a todos los países la OCDE (Organización para la Cooperación y el Desarrollo Económico) tienen un sistema de comercialización que los estados miembros que lo deseen pueden aplicar voluntariamente. Según estas normas el material de base está constituido por las poblaciones, plantaciones y clones de los que se obtiene el material forestal de reproducción. Los diferentes tipos de materiales de base que están admitidos para las especies reguladas por estas normas son: fuentes semilleras, rodales, huertos semilleros, progenitores de familia, clones y mezcla de clones. Se establecen cuatro categorías de material forestal de reproducción: identificada, seleccionada, cualificada y controlada, que pueden proceder de distintos tipos de materiales de base (Tabla 3) y los cuales son equivalentes a las categorías establecidas por la normativa chilena (ver detalles más adelante en el texto). |
||
 |
||
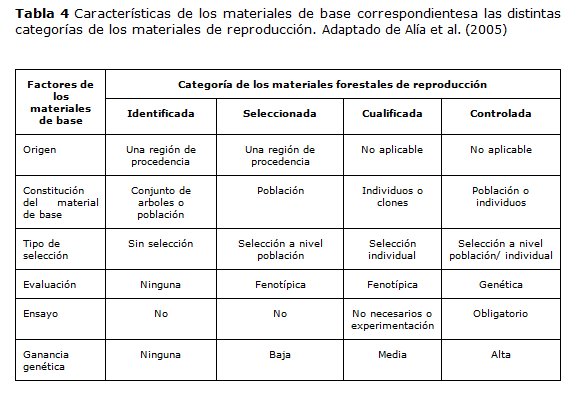
| De acuerdo a su categoría y al material de base del que proceden, los materiales de reproducción presentan características diferentes en cuanto a origen, componentes, tipo de selección y evaluación (Tabla 4), que están caracterizados por: i) la variación y diferenciación genética del material de base; ii) el tipo e intensidad de selección aplicado; iii) el esquema de cruzamiento utilizado para obtener los materiales de base; iv) el método de propagación y esquema de cruzamiento utilizado para obtener los materiales de reproducción; v) el tipo de ensayos necesarios para evaluarlos; vi) la ganancia genética obtenida. | ||
 |
||
En Chile existe una autoridad nacional responsable de la certificación de semillas (SAG), una ley (DL N° 1.764) y reglamentos complementarios que permiten su implementación en semillas de cultivo agrícolas y frutales, pero se carece de reglamentación específica para especies forestales. Hasta el año 2006 no existieron estándares oficiales de calidad para plantas de especies forestales. Ese año el Instituto Nacional de Normalización (INN) junto al INFOR, el SAG y otros participantes, crean la Norma Chilena Oficial 2957 que regula el proceso de certificación de la producción y comercialización de materiales de propagación de uso forestal para las especies P. radiata, E. globulus, E. nitens, Pseudotsuga menziesii y N. dombeyi (INN 2006). Con esta norma, la idea de certificación aparece por primera vez en el mercado de plantas forestales, integrándose como el primer eslabón en la cadena de custodia (trazabilidad). Esta norma, de carácter voluntario, se fundamenta en la necesidad de estándares de calidad para plantas forestales, estableciendo los requisitos morfológicos y fisiológicos mínimos que debe cumplir cada especie para obtener la certificación. Sin embargo, aun se carece de pautas básicas de ordenamiento genético territorial lo que podría estar ocasionando pérdidas económicas por uso de materiales que no están adaptados y pérdidas de diversidad genética en procedencias locales, debido a contaminación producida por individuos de otras procedencias. Conscientes de esta situación, el INFOR como beneficiario, y el SAG como mandante y oferente, ejecutan actualmente un proyecto INNOVACHILE, con el objetivo de "Desarrollar una Propuesta de Reglamento de Semillas y Plantas Forestales", para reducir la incertidumbre que al momento de la compra enfrentan los forestadores respecto a la idoneidad y desempeño futuro de estos materiales. Para tal efecto se propone desarrollar normativas técnicas que permitan regular las áreas de producción y utilización de semillas (mapa de regiones de procedencia y establecer estándares de calidad para las plantas), y articularlas en un Reglamento de Certificación de Semillas y Plantas Forestales, el cual se generará mediante un proceso amplio de consulta a expertos y a los actores relevantes involucrados en esta materia.
Regiones de procedencia En el caso de especies forestales, se trata de buscar las semillas que contengan las variantes genéticas (o alelos) relacionadas con caracteres de crecimiento, producción y adaptación, más idóneas para una determinada ubicación geográfica. Para ello se seleccionan grandes unidades territoriales para la comercialización de materiales de reproducción según su procedencia, obteniéndose las semillas de rodales, huertos o de árboles padres selectos. Las fuentes semilleras están constituidas por árboles situados dentro de una zona de recolección de frutos y semillas, con una superficie tal que contenga uno o más grupos de árboles bien distribuidos y en número y densidad suficiente para asegurar una adecuada polinización cruzada. La utilización de una fuente de semilla inadecuada puede producir la pérdida parcial o total de la reforestación, pero el peor daño que se puede producir es que la masa instalada logre llegar a la edad de reproducción y su polen entre en competencia con masas naturales de la zona (Zobel y Talbert, 1984; Mesén et al., 1996). Para las especies a las que se aplica un sistema de certificación de materiales de reproducción, se definen unidades de comercialización en los materiales identificados y seleccionados (Tabla 3 y 4), que corresponden a divisiones de su distribución geográfica. A cada unidad básica resultante de la división en regiones se la denomina región de procedencia. La región de procedencia se define para una especie como la zona o el grupo de zonas sujetas a condiciones ecológicas suficientemente uniformes en las que se encuentran fuentes semilleras o huertos que presentan características fenotípicas o genéticas semejantes, teniendo en cuenta límites de altitud, cuando proceda (Zobel y Talbert, 1984). La región de procedencia determina los límites geográficos máximos dentro de los cuales se puede mezclar la semilla recolectada de fuentes semilleras. Según la normativa internacional de la OCDE (2007) si no se han establecido regiones de procedencia, por no tener fuentes semilleras o huertos identificados que justifiquen la producción de materiales de reproducción, no se puede comercializar material de reproducción de las categorías identificado o seleccionado a partir de materiales de base. Solo podrían aprobarse materiales de base para la producción de materiales de reproducción de las categorías cualificada y controlada (Tabla 3 y 4), lo cual demanda más tiempo, ensayos y recursos para su aprobación. Para la delimitación de regiones de procedencia dos son los métodos más utilizados: i) aglomerativo y ii) divisivo (CTGREF, 1976; Lindgren y Ying, 2000; Kleinschmit et al., 2004; Geburek y Konrad, 2008). El método aglomerativo se basa en la unión en una misma región de procedencia de las zonas (o fuentes semilleras o rodales) de una especie que presentan características ecológicas, fenotípicas o genéticas similares. Las zonas homogéneas de cada especie se determinan tras analizar los patrones de variación fenotípicos y genéticos, así como los factores que más influyen en la diferenciación de las poblaciones: aislamiento geográfico, características ecológicas, etc. Con respecto al método divisivo, se realiza una división territorial en zonas con características ecológicas similares, que se supone darán lugar a fuentes semilleras o rodales con características fenotípicas o genéticas similares. La división se suele realizar a partir de las regiones biogeoclimáticas. Muchas veces el interés diferencial por las especies, así como el distinto grado de conocimiento y de los factores que ocasionan su variación genética, obliga a que se usen los dos métodos. Los criterios para establecer esas uniones o divisiones territoriales son, por un lado la similitud ecológica, y por otro la similitud en las características fenotípicas o genéticas de las masas forestales que habitan en ellas. La aplicación práctica difiere entre los distintos países, pues las regiones establecidas dependen de la información disponible sobre las especies o el territorio. En general se utilizan distintos criterios, tales como ecológicos (clima, geología, suelo), geográficos, de distribución de las especies, económico-silvícolas y relacionados con límites administrativos. La información disponible sobre la variación genética de las especies se utiliza en pocos casos, ya que esta información suele ser incompleta y referida a pocas poblaciones. En el mejor de los casos los datos procedentes de los ensayos de procedencias-progenies y de marcadores moleculares pueden ser utilizados para precisar los límites entre las regiones (Alía et al., 2005).
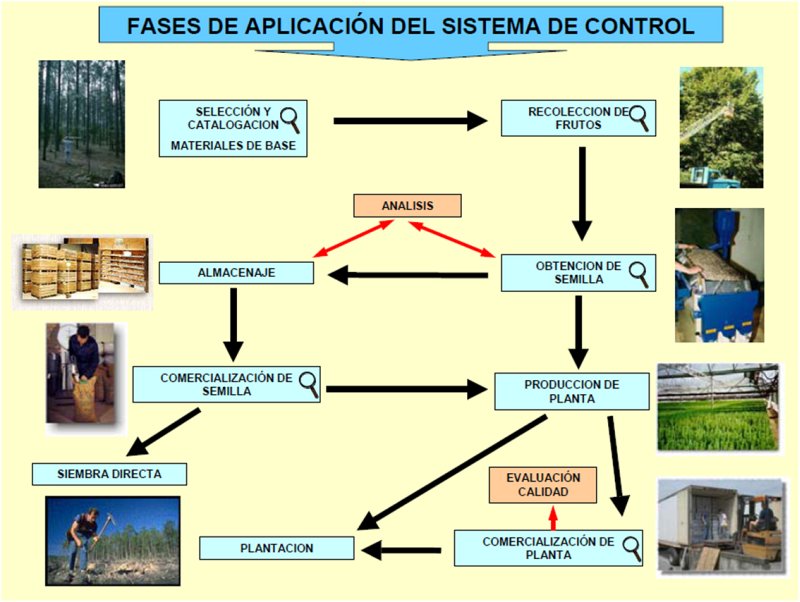
Control de los materiales forestales de reproducción El sistema de control consta de dos componentes fundamentales; 1) una serie de normas y 2) una organización administrativa. Esta última, en base a las normas monitorea los procesos de producción y comercialización, asegurando que la identidad (origen) y la calidad genética (categoría) asignada a un determinado material forestal de reproducción no sea alterado en ninguna fase del proceso, y garantiza que su calidad exterior cumple con valores dentro de rangos establecidos. Su establecimiento se basa en la necesidad de protección del usuario de materiales forestales ante la práctica imposibilidad de reconocer de manera objetiva e inmediata la identidad y de evaluar la calidad genética del material de reproducción. La correcta aplicación de la normativa necesita el apoyo de herramientas y procedimientos específicos para el control de su cumplimiento. En este sentido, las técnicas de marcadores moleculares se perfilan como instrumentos adecuados de diagnóstico, ya que permiten a productores como a controladores, un apropiado seguimiento en los procesos de producción, comercialización y utilización de algunos tipos de material forestal de reproducción (Figura 1). Con un sistema de control bien establecido basado en marcadores moleculares se puede conseguir la trazabilidad completa desde el material base, pasando por la fase de semilla, planta, materia prima sin procesar (productos madereros y no madereros) hasta el producto final (Finkeldey et al., 2010). |
||
Figura 1 Representación esquemática de la cadena productiva de plantas forestales indicando los momentos más adecuados para control visual o diagnóstico molecular. |
||
 |
||
Actualmente se dispone de un amplio número de marcadores moleculares, herramientas que están en constante aumento y perfeccionamiento. Dichas herramientas tienen diversas potencialidades para el diagnóstico, de modo que puede afirmarse que existe un marcador para cada objetivo, como por ejemplo; i) caracterización rápida de los niveles de diversidad genética de las poblaciones (e.g. isoenzimas, RAPD), ii) identificación y verificación de la autenticidad del material forestal de reproducción (e.g. barcodes, SSR, SCAR), iii) estimación simple de otros parámetros genéticos relacionados con su calidad como la heredabilidad (e.g. SSR, AFLP, SNPs). El uso de marcadores moleculares en el proceso de control de los materiales de base y de reproducción está bien demostrado y documentado (Alía et al. 2005, Deguilloux et al. 2003, Deguilloux et al. 2004, Finkeldey et al. 2010), al igual que su uso en el género Nothofagus (Mattioni et al. 2002, Azpilicueta et al. 2004, Premoli y Kitzberger 2005, Gallo et al. 2006, Marchelli et al. 2008, Mathiasen y Premoli. 2010). A nivel práctico, los marcadores moleculares tienen diversas aplicaciones en el campo de la conservación y el mejoramiento genético (Glaubitz y Moran 2000): – Medida de la diversidad genética y la diferenciación – Estimación de las tasas de flujo genético o migración – Caracterización del sistema de reproducción – Análisis de paternidad y parentesco – Determinación de la eficiencia de los huertos semilleros – Identificación de clones y material forestal de reproducción – Estudios de filogenia y taxonomía – Mapas de ligamiento genético, análisis de genes que controlen rasgos cuantitativos (QTLs), selección asistida por marcadores. En este punto es necesario resaltar que así como los marcadores moleculares tienen sus ventajas, también tienen desventajas que pueden limitar su aplicabilidad. Las diferencias entre poblaciones de una misma especie que ocupan distintas regiones se pueden atribuir, en parte, a procesos de modificación fenotípica como respuesta a las distintas condiciones climáticas y edáficas locales. Sin embargo, existen importantes diferencias que tienen una base genética y han sido objeto de selección natural o artificial a lo largo de un determinado número de generaciones. Estas diferencias genéticas son la base sobre la que se establece la delimitación de las regiones de procedencia de las principales especies forestales, como unidad básica de comercialización en los materiales de reproducción. Se distinguen dos tipos de variación; la diversidad neutral, es decir, aquellos rasgos no determinados por fuerzas selectivas, y la variación adaptativa, constituida por los caracteres con valor adaptativo. La primera se estima principalmente mediante marcadores moleculares, existiendo diversas técnicas para estudiar tanto proteínas como el ADN. El método clásico de estudio de la variación adaptativa son los ensayos de procedencia/progenies, en los que se analiza el grado de variabilidad y el porcentaje de ésta que corresponde a una variación genética (Jiménez y Collada 2000). La base de estos estudios es ensayar distintos genotipos en un mismo ambiente con el fin de minimizar la variabilidad ambiental (Donoso y Gallo 2004). Sin embargo, la presencia de alelos únicos o que muestran diferencias significativas en su frecuencia para distintos grupos (detectados mediante marcadores moleculares), puede ser un método efectivo de estudiar y utilizar la variación adaptativa.
Variación intraespecífica en Coigüe En el caso concreto de Coigüe por su amplia distribución geográfica (en Chile crece entre las provincias de Colchagua en Región de O'Higgins y Capitán Prat en Región de Aysén, en ambas cordilleras y el sur de Argentina), con poblaciones grandes y continuas debería esperarse una elevada variación genética poblacional y un elevado flujo génico interpoblacional que reduce la divergencia interpoblacional, como fue demostrado mediante estudio de isoenzimas (Donoso et al. 2004, Donoso et al. 2006). La mayoría de la variación genética se encontró dentro de las poblaciones, mientras que el porcentaje entre distintas poblaciones resultó ser muy bajo. Del total de loci, el 70% resultó monomórfico en al menos una población. La heterocigosis observada (Ho) varió entre 0,063-0.104 y la heterocigosis esperada (He) entre 0,059-0,130 (Premoli 1996). No obstante, se debe tener en cuenta que la variación electroforética detectable en isoenzimas es una pequeña fracción de la diversidad genética real de la población. Por otra parte, dichas herramientas moleculares complican la búsqueda de alelos únicos que tengan el potencial de ser utilizados en programas de mejoramiento y a la vez justifican el uso de marcadores moleculares altamente informativos y con buena capacidad de multiplexación (análisis de varios marcadores en una misma reacción). Entre estos marcadores se encuentran los SSRs, AFLP, y los SNP (Single Nucleotide Polymorphism). Merchelli et al. (2008) y Soliani et al. (2010) desarrollan SSRs para Raulí y Lenga, respectivamente. Estos marcadores demuestran ser altamente polimórficos y a la vez transferibles a otras especies de Nothofagus como el Coigüe, detectándose heterocigocidades entre 0,604-0.834 y 0,118-0,867. Los AFLP ya fueron probados en N. alpina mostrando tasas de polimorfismo > 50% y heterocigocidades > 0.7 (Hasbún et al. 2011). Por su parte los SNP altamente polimórficos por naturaleza, no han sido probados en el género Nothofagus han comenzado a ser asequibles gracias a las nuevas tecnologías de secuenciación y genotipificación que han reducido notable su costo de uso mediante nuevas tecnologías como "Genotyping by Sequencing" (GBS) (Elshire et al. 2011). En el sur de Asía las distintas especies milenarias de Shorea (muy apreciadas por su madera) se han visto favorecidas con la aplicación de los marcadores AFLP, los cuales han ayudado a identificar especies, procedencias y genotipos locales y no locales, determinar zonas en las que se está perdiendo diversidad y facilitar la introducción de nueva diversidad para los programas de conservación integrada y de mejoramiento genético (Cao et al. 2006, Nuroniah 2009, Finkeldey et al. 2010). Esta herramienta puede servir también para mejorar el manejo de la variación genética intraespecífica (Brown y Kresovich 1996, Karp 2000). Por otra parte los SNPs son ampliamente utilizados en especies agrícolas y frutales para clasificación de germoplasmas. Las frecuencias alélicas en diferentes colecciones de germoplasma son estimadas y utilizadas para identificar marcadores SNP con alelos únicos o alelos perdidos en colecciones de germoplasma específicos. Los alelos únicos por colección indican los alelos existentes sólo en un banco de germoplasma, pero no en los demás. Los alelos perdidos indican los alelos ausentes por completo en una colección de germoplasma, mientras que están presentes en todos las otras (Lu et al. 2009). Este tipo de marcadores comienzan a ser una opción real de utilización en especies forestales mediante GBS, tecnología que ya fue probada en eucalipto (Faria el al., 2011).
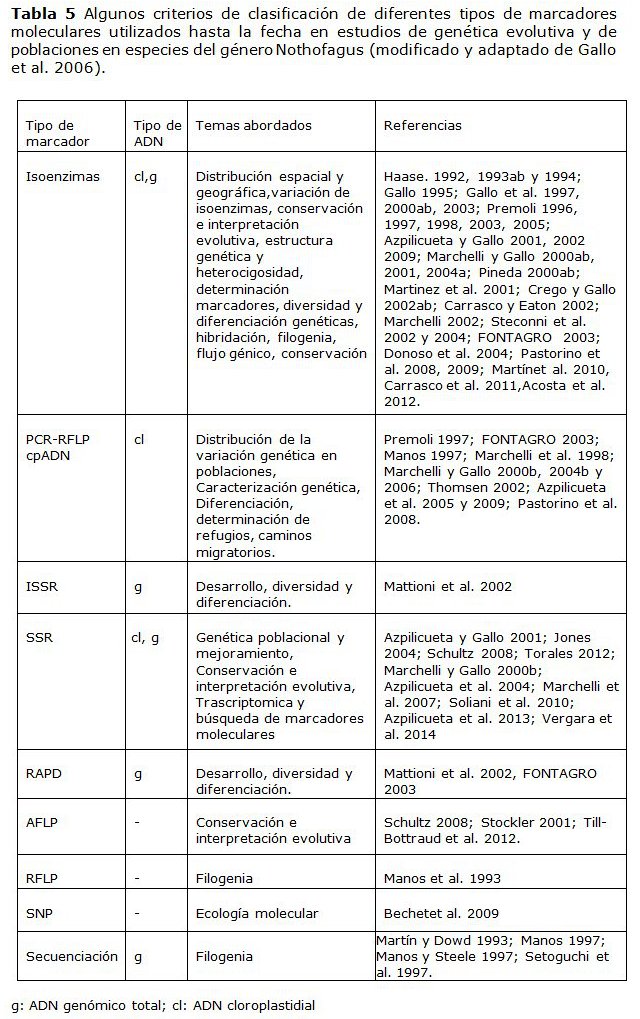
Marcadores moleculares en el género Nothofagus En relación a los bosques chilenos y el manejo de nuevas tecnologías aún existe mucho por investigar por ejemplo el género Nothofagus (N. alessandrii, N. alpina, N. antartica, N. betuloides, N.dombelyi, N. glauca, N. nítida, N. obliqua , N. pumillo) especies que crecen al Sur de Chile y Argentina todavía se desconoce mucho en lo respecta el área de genética y herramientas moleculares, aunque los estudios comenzaron hace alrededor de 20 años solo se tiene referencia de datos de aproximadamente 10 años atrás, los cuales no logran entregar toda la información necesaria para ir solucionando problemas latentes, careciendo de un genoma secuenciado las especies chilenas de este género resultan muy difícil solucionar los actuales problemas de selección, conservación y mejoramiento genético. Los primeros trabajos con marcadores moleculares en especies del genero Nothofagus corresponden a estudios de taxonomía y clasificación filogenética (Haase 1992), estudios que fueron cerciorados en el año 2006 mediante la publicación de Gallo et al. (2006). Una exhaustiva revisión bibliográfica en diferentes bases de datos arrojó que principalmente se han utilizado marcadores isoenzimatico, los cuales ya representan solo una porción menor de la diversidad genética, es decir son pocos informativos. Otros estudios realizados corresponden a caracterización de poblaciones (FONTAGRO 2003, Gallo et al. 2006) pero estos solamente abocados mayoritariamente aespecies N. obliqua y N. alpina (Gallo et al.2006, Azpilicueta et al.2009). En Argentina un proyecto puesto en marcha "Estudio de genes candidatos para estrés abiótico en especies forestales nativas patagónicas", proyecto a cargo del Área Estratégica Biología Molecular, Bioinformática y Genética de Avanzada y del Programa Nacional Forestal del INTA, investigadores del Instituto de Recursos Biológicos, Instituto de Biotecnología y la EEA Bariloche, tuvo por objetivo desarrollar y analizar el genoma funcional de hoja de N. nervosa, un recurso genético forestal nativo patagónico de gran interés para la región por su valor económico y ecológico (INTA. 2013). En el proyecto se descubrió un número considerable de SSR los cuales fueron registrados. Para confirmar la validez de estos marcadores se seleccionó un subgrupo de 73 SSR los cuales mediante pruebas funcionales fueron amplificados con éxito en seis muestras de ADN deplántulas, siendo 14 muy polimórficos. Por último, también se ha analizado la variación con marcadores en ADN de cloroplasto, que por sus características (tasa de evolución altamente conservada y herencia uniparental (materna) y clonal) permiten estudiar la historia evolutiva de las especies. Además, en este caso se estudiaron 15 poblaciones de Chile. Con estos marcadores se observaron diferencias entre las Cordilleras de la Costa y de los Andes y, en estaúltima, dos grandes grupos de poblaciones separadas en sentido norte-sur. |
||
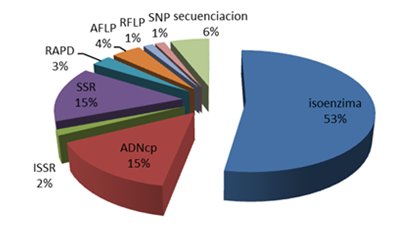
| Gráfico 1. Número de publicaciones en porcentaje, según tipo de marcador molecular, entre los años 1992-2014, sin considerar estudios en progreso. | ||
 |
||
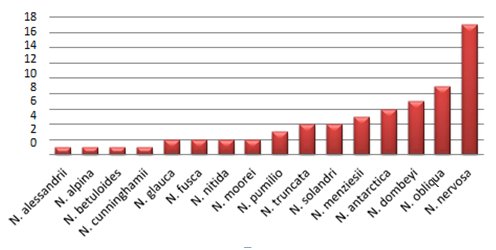
| Gráfico 2. Número de estudios por especies del genero Nothofagus (nativas y exóticas), en relación al uso de marcadores moleculares (más detalle en la tabla 5). | ||
 |
||
 |
||
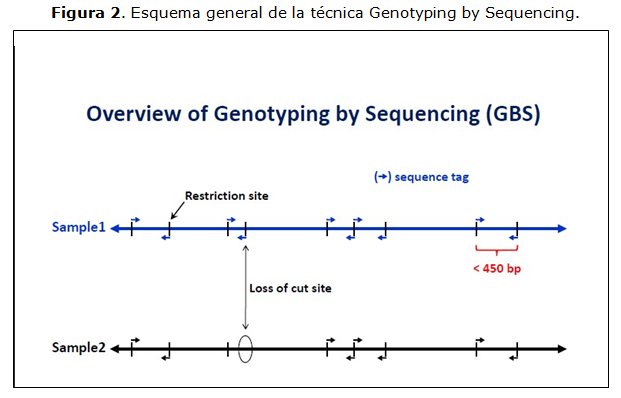
Nuevas herramientas moleculares basadas en enzimas de restricción y secuenciación de alto rendimiento El descubrimiento de las enzimas de restricción permitieron el desarrollo de los primeros marcadores moleculares (RFLP) y han ido contribuyendo de manera continua a gran parte de las nuevas herramientas que van surgiendo (ej. AFLP, SNP). Las enzimas de restricción son fundamentales para la genotificación de alto rendimiento basada en secuenciación de nueva generación (NGS; Next Generation Sequencing). Hoy en día estas dos técnicas han ido evolucionado al punto que su fusión dio origen a la técnica de genotipificación por secuenciación (GBS; Genotyping by Sequencing), donde los polimorfismos en lugar de estar basados en la longitud de los fragmentos, se buscan en variantes estructurales o SNP (Elshire et al. 2011). La diversidad de enzimas de restricción disponibles (que varían en la longitud, sitios de reconocimiento, y también en su sensibilidad a metilación) permite optimizar el método a cualquier especie. Su flexibilidad permite a los investigadores personalizar enfoques de investigación y descubrimiento de marcadores en proyectos individuales (Davey 2011). La genotipificación por GBS es un sistema sencillo, robusto y eficiente para la construcción de librerías, basándose en la digestión del ADN para una reducción representativa del genoma y la determinación de la secuencia de los fragmentos basado en técnicas de secuenciación masiva en sistema tales como Illumina, 454, Ion torrent, etc. El enfoque de ésta técnica es genotipificación por secuenciación y es adecuada para todo tipo de genomas, incluso aquellos grandes y complejos, ya que es capaz de generar una versión reducida de este, mediante el uso de enzimas de restricción. En particular se usan enzimas de restricción altamente sensibles a la metilación, lo que permite que grandes bloques de ADN altamente metilados (normalmente asociados a zonas repetitivas) queden fuera del análisis. De esta forma se cubren zonas genómicas codificantes y no codificantes que comúnmente quedan fuera con otras plataformas (ej. RNA-seq). Si a esto le agregamos la utilización de un eficiente sistema de multiplexación basado en el uso de "etiquetas" o barcodes que permite analizar en paralelo cientos de muestras, convierte a GBS en una técnica de alto rendimiento (Craig et al. 2008). La técnica fue desarrolla con el objetivo de crear un protocolo barato y solido de secuenciación múltiple (Elshire et al. 2011). Sin duda ha sido una de las plataformas más convenientes que han surgido en relación a la genotipificación y descubrimiento de SNP, ya que permite ambos procesos de una forma eficiente y a un bajo costo. Es una tecnología nueva que está siendo ocupada para diversos objetivos como un nuevo enfoque de genotipificado en especies que no tengan un genoma de referencia, o en especies polipoloides como el trigo. También se usa para la construcción de mapas genéticos de alta densidad, caracterizar germoplasmas y desarrollar modelos de selección genómica para programas de mejoramiento. Esta técnica como se mencionaba anteriormente genera grandes cantidades de marcadores SNP (Elshire et al. 2011, Lu et al. 2013, Nimmakayala et al. 2014), basados en los sitios de reconocimiento de las enzimas, los cuales al variar una base estos se pierden contribuyéndonos un polimorfismo (Figura 2). |
||
 |
||
Los marcadores obtenidos se pueden usar para análisis genéticos incluyendo estudios de población, caracterización de germoplasmas y mapeo de algunos caracteres en diversos organismos etc., y en aplicaciones futuras ya que permitirá a los cultivadores y mejoradores genéticos la selección de germoplasmas elite de especies de interés, sin realizar estudios moleculares previos o bien para hacer planes de conservación sin tener estudios genéticos previos. Dentro delas ventajas deesta técnica cabemencionar las siguientes: - Reducción en la manipulación de la muestra - Pocos pasos en PCR y purificación - Tamaño DNA pequeño (fraccionado) - Eficiente sistema de bar-codes - Búsqueda y genotipificación simultánea Esta técnica depende y requiere de ADN de excelente calidad y alta pureza (libre de ARN). Actualmente se utiliza solamente 100 ng, aunque se están probando disminuir a 50 ng, pero en estricto rigor se necesita 1uL de muestra. Regularmente los laboratorios que prestan servicio piden al menos 1ug de ADN por muestra, porque necesitan ADN extra para la robótica, repeticiones ocasionales y porque los usuarios suelen sobrestimar la concentración de su ADN. |
||
Cambio climático en Chile y particularidades de Chile centro-sur No se puede dejar de considerar que el clima constituye uno de los principales factores que dirigen los patrones de distribución espacialy productividad de las plantas. Las variables componentes del clima, se relacionan con los rangos geográficos y ecológicos de distribución de las plantas, de acuerdo a las restricciones fisiológicas que éstas presentan y determinan la supervivencia y productividad de sus poblaciones en un determinado lugar (Woodward 1987, Lomolino et al. 2006). Existe amplia evidencia acerca de la naturaleza dinámica del clima (Petit et al. 1999, Hinojosa 2005), la cual impulsa a las especies a ir adaptándose a las condiciones cambiantes (Woodward 1987, Villagrán & Hinojosa 1997). Pese a esto, la tasa de cambios del clima durante las últimas décadas, ha sido muy superior al ritmo de los siglos anteriores (Gitay et al. 2002, IPCC 2007). Una variable clave como la temperatura media global, ha presentado un aumento de cerca de 1°C durante el último siglo (IPCC 2007). Si la tasa de cambios en el clima excede el ritmo de respuesta fisiológica de las especies, en especial la capacidad de las poblaciones de migrar o adaptarse, se puede esperar un profundo impacto adverso en la distribución de las especies, estructuras de comunidades y procesos ecosistémicos asociados (Ackerly et al. 2010). Las modificaciones que el cambio climático ocasiona en la distribución de las plantas y disminución de sus tamaños poblacionales, se asocian al hecho que los hábitats de muchas especies se desplazarán hacia los polos o hacia mayores altitudes desde sus localidades actuales (Walther et al. 2002, Lawler et al. 2006, Lenoir et al. 2008, Ackerly et al. 2010). La tendencia de cambio climático para Chile centro-sur observa una merma de entre un 20-25% de precipitación anual registrada en las últimas décadas, y se prevé que continúe en el futuro (CONAMA 1999, CONAMA 2008, Searle & Rovira 2008). A ello debe sumarse el que una disminución significativa en el monto de las precipitaciones, generalmente va asociada a una mayor variabilidad en la periodicidad de las mismas (Woodward 1987). Por otra parte, la tendencia global de aumento en temperatura media anual también sería aplicable a las regiones centrales y sureñas de Chile, lo que conjuntamente podrá reducir aún más el régimen hídrico en las cuencas y por ende su disponibilidad para los ecosistemas y su productividad en estas regiones (CONAMA 2008, Searle & Rovira 2008). |
||
Hipótesis del Proyecto Existen herramientas modernas de tipo moleculares y de modelamiento ecológico que pueden ayudar a definir de forma eficiente y con proyección a largo plazo, regiones de procedencia de materiales de propagación de coigüe en Chile junto con permitir la certificación de origen. |
Métodos Existen herramientas modernas de tipo moleculares y de modelamiento ecológico que pueden ayudar a definir de forma eficiente y con proyección a largo plazo, regiones de procedencia de materiales de propagación de coigüe en Chile junto con permitir la certificación de origen. |
Bibliografía Ackerly D, Loarie S, Cornwell W, Weiss S, Hamilton H, Branciforte R, Kraft N. 2010. The geography of climate change: implications for conservation biogeography. Diversity and Distributions 16(3):476-487. Acosta M., Mathiasen P., Premoli A. 2002. Predominant regeneration strategy results in species-speci?c genetic patterns in sympatric Nothofagus s.s. congeners (Nothofagaceae). Australian Journal of Botany 60:319–327. Alía R., Alba N, Agúndez D, Iglesias S. 2005. Manual para la comercialización y producción de semillas y plantas forestales. Materiales de base y de reproducción. Serie Forestal. DGB. Madrid. 384 pp. Azpilicueta M., Marchelli P., Gallo L. 2009. The effects of Quaternary glaciations in Patagonia as evidenced by chloroplast DNA phylogeography of Southern beech Nothofagus obliqua. Tree Genetics & Genomes5:561–571. Azpilicueta M., Gallo L.2001. Análisis de la variación genética en Nothofagus obliqua (Mirb.) Oerst. a través de marcadores génicos isoenzimáticos. In XXX Congreso Argentino de Genética, Mar del Plata, Argentina. Actas. p. 147. Azpilicueta M., Gallo, L., 2002. Variación genética aloenzimatica como herramienta de diagnóstico para la conservación e interpretación evolutiva de los bosques de Nothofagus obliqua (Mirb.) Oerst. en Argentina. In Actas XXXI Congreso Argentino de Genética, La Plata. Actas. p. 135. Azpilicueta M., Caron H., Bodénès B., Gallo L. 2004. SSR markers for analysing South American Nothofagus species. Silvae Genetica 53: 240-243. Azpilicueta M., Marchelli P., Gallo L. 2005. Common ice-age refugia in two southernbeeches (Nothofagus spp.). In XXII IUFRO Congress “Forest in the balance: linking tradition and technology”, Brisbane, Australia. p 68. Azpilicueta M., Gallo L.,Van zonneveld M., Thom E., Moreno, C.Marchelli P. 2013. Management of Nothofagus genetic resource: Definition of genetic zones based on a combination of nuclear and chloroplast marker data. Forest Ecology and Management 302:414-424. Barret CW, JF Jacobs. 2006. Quality assessment of temperate zone deciduous hardwood seedlings. New Forests 31:417-433. BechetA.,Varios.2009. Permanent Genetic Resources added to Molecular Ecology Resources Database 1 October 2009–30 November. Brown S, Kresovich S. 1996. Molecular characterization for plant genetic resources conservation. En: A.H. Paterson (Ed.) Genomic mapping in plants. R.G. Landes Austin, TX, USA. pp. 85–93. CarrascoB., EatonL. 2002. Natural history and genetic structure of Raulí (Nothofagus nervosa (Phil.) Dim. et Mil.). Forest Genetics 9:275-284. Carrasco B., Eaton L.,Letelier L., Díaz C., García-Gonzáles R. 2011. Heterogeneousgeneticstructureina natural population of Raulí (Nothofagus nervosa). Ciencia eInvestigación Agraria. 38:441-452. Cao C-P, Finkeldey R, Siregar I, Siregar U, Gailing O. 2006. Genetic diversity within and among populations of Shorea leprosula Miq.and S. parvifolia Dyer (Dipterocarpaceae) in Indonesia detected by AFLPs. Tree Genet Genomes 2:225–239. Castillo J, Moreno G. 2002. Semillas forestales del bosque nativo chileno. Editorial Universitaria. Santiago, Chile.223 pp. CONAF. 2005. Viveros forestales, temporada 2004-2005. Chile Forestal 314. 24 p. CONAF. 2006. Viveros forestales, temporada 2006. Chile Forestal 322. 28 p. CONAMA. 1999. Primera Comunicación Nacional bajo la Convención Marco de las naciones Unidas sobre el Cambio Climático. Comisión Nacional del Medio Ambiente. Santiago, Chile. 153 pp. CONAMA. 2008. Plan de Acción Nacional de Cambio Climático 2008-2012. Comisión Nacional del Medio Ambiente. Santiago, Chile. 86 pp. Craig D., Pearson J., Szelinger S., Sekar A., Margot R., Corneveaux J., Pawlowski T., Laub T., Nunn G., Stephan D.,Homer N., Huentelman M. 2008. Identification of genetic variants using barcoded multiplexed sequencing. Natural methods .5(10): 887–893. Crego, M., Gallo L. 2002a.Local dynamic of interspecific hybridization between two southern beeches (Nothofagus spp.). In DYGEN Conference. “Dynamics and conservation of genetic diversity in forest ecosystems”. Strasbourg, France. Abstracts. p. 108. CregoM., Gallo L., 2002b.Variación temporal en la hibridación natural entre Nothofagus nervosa, Raulí y Nothofagus obliqua, Roble Pellín. In XXXI Congreso Argentino de Genética. La Plata. Actas. p.136. CTGREF. 1976. Semences Forestieres. Les regions de provenance d’epicea commun. CTGREF Nogent/Vernison. Not. Tech. Num 30. Davey J.,Hohenlohe P.,EtterP.,BooneJ.,CatchenJ.,MarkL.,2011. Genome-wide genetic marker discovery and genotyping using next-generation sequencing.Nature Reviews Genetics 12:499-510. Deguilloux MF, Pemonge MH, Bertel L, Kremer A, Petit RJ. 2003. Checking the geographical origin of oak wood: molecular and statistical tools. Molecular Ecology 12:1629-1636. Deguilloux MF, Pemonge MH, Petit RJ. 2004. DNA-based control of oak wood geographic origin in the context of the cooperage industry. Annals of Forest Science61: 97-104. Donoso C, A Premoli, PJ Donoso. 2004. Variación genética en Nothofagus siempreverdes (Coihues). En: Donoso C, Premoli A, Gallo L, Ipinza R (Eds.) Variación intraespecífica en las especies arbóreas nativas de los bosques templados de Chile y Argentina. Editorial Universitaria,Santiago, Chile. pp. 189-211. Donoso C, Gallo L. 2004. Aspectos conceptuales y metodológicos. En: Donoso C (Ed.) Variación intraespecífica en las especies arbóreas nativas de los bosques templados de Chile y Argentina. Editorial Universitaria,Santiago, Chile. pp. 189-211. Donoso P, Soto D. 2010. Plantaciones con especies nativas en el centro-sur de Chile: experiencias, desafíos y oportunidades. Revista Bosque Nativo 47:10-17. Donoso PJ, C Donoso, B Escobar, C Navarro, L Gallo. 2006. Nothofagus dombeyi. En: Donoso C (Ed.) Especies arbóreas de los bosques templados de Chile y Argentina. Autoecología. Marisa Cúneo Ediciones, Valdivia, Chile. pp. 423-432. Donoso PJ, González ME, Escobar B, Basso I, Otero L. 1999. Viverización y plantaciones de Raulí, Roble y Coihue. En: C. Donoso y A. Lara (Eds.) Silvicultura de los Bosques Nativos de Chile. ED. Universitaria, Santiago, Chile. pp. 177-244. Donoso C., Gallo L., Donoso P., Azpilicueta M. 2004. Variación en Nothofagus obliqua (Mirb.). Variación intraespecífica en las especies arbóreas de losbosquestempladosdeChiley Argentina. Santiago de Chile, Chile, Editorial Universitaria. p. 79-113. Elshire R. ,Glaubitz J., Poland J., Kawamoto K.,Buckler E., MitchellS. .2011. A Robust, Simple Genotyping-by-Sequencing (GBS) Approach for High Diversity Species. PLoS ONE 6(5): e19379. doi:10.1371/journal.pone.0019379. Faria D,Tanno P, Reis A, Martins A, Ferreira M, Grattapaglia D. 2011. Genotyping-by-Sequencing (GbS) the Highly Heterozygous Genome of Eucalyptus Provides Large Numbers of High Quality Genome-Wide SNPs. PAG XX, San Diego, CA, USA. http://pag.confex.com/pag/xx/webprogram/Paper4239.html. Finkeldey R, Leinemann L, Gailing O. 2010. Molecular genetic tools to infer the origin of forest plants and wood. Appl Microbiol Biotechnol 85: 1251-1258. FONTAGRO. 2003. Caracterización genética de poblaciones de Nothofagus obliqua y Nothofagus alpina mediante marcadores moleculares e isoenzimatico”, Chile- Argentina. Gallo L. 1995. Supuestos híbridos interespecíficos en progenies de Raulí. Resumen. In Congreso Argentino de Genética. 26. Reunión Anual de la Sociedad Genética de Chile. 28. Jornada Argentino-Chilena de Genética. 1. 1995 10 22-26, 22-26 de octubre, 1995. San Carlos de Bariloche. Gallo L., Marchelli P., Breitembücher A. 1997. Morphologycal and allozymic evidence of natural hybridization between twosouthernbeeches(Nothofagusspp.) and its relation to heterozygosity and height growth.Forest Genetics 4: 13-21. Gallo LA, Marchelli P, P Crego. 2000b. Variación genética en poblaciones y progenies de Raulí en Argentina. III. Dinámica de hibridación interespecífica entre Raulí y Roble Pellín. In R Ipinza, B Gutiérrez y V Emhart eds. Domesticación y Mejora Genética de raulí y roble. Valdivia, Chile. Exsion. p. 181-196. Gallo L.,Marchelli P., Crego P., Oudloerlo L., Izquierdo F., Breilembücher A., González M., Chauchard L., Maresea L., Cuerpo de Guardaparques de los Parques Nacionales Lanin & Nahuel Huapi., Mel U. 2000b. Revista Domesticación y mejora genética en Rauli y Roble.Editorial Maval Ltda.ISBN: 956 288-691-3 Registro de Propiedad IntelectualN°115.794 Agosto 2000. Páginas 157-180. Gallo L., Marchelli P., González Peñalba M., Chauchard L., & Vellido S. 2003. Genética de poblaciones en la definición de áreas protegidas. In Simposio de Recursos Genéticos para América Latina y el Caribe. Gallo LA, P Marchelli, MM Azpilicueta, P Crego. 2006. El uso de marcadores genéticos en el género Nothofagus con especial referencia a raulí y roble. Bosque 27:3-15. Geburek T, Konrad H. 2008. Why the conservation of forest genetic resources has not worked. Conservation Biology 22:267-274. Gitay H, Suárez A, Watson R, Dokken D. 2002. Climate change and biodiversity. IPCC. Intergovernmental Panel on Climate Change. 86 pp. Glaubitz J, Moran G. 2000. Genetic tools: the use of biochemical and molecular markers. En: Young A, Boshier D, Boyle T (Eds.). Forest conservation genetics: principles and practice. CSIRO-CABI, pp. 39-59. Haase, P. 1992. Isozyme variability and biogeography of Nothofagus truncata (Fagaceae). New Zealand Journal of Botany 30: 315-328. Haase, P. 1993a. Isozyme studies of New Zealand Nothofagus species (southern beech) using leaf extracts. Silvae Genetica 42: 46-51. Haase, P. 1993b. Genetic variation, gene flow, and the “founder effect” in pioneer populations of Nothofagus menziesii (Fagaceae), South Island, New Zealand. Journal of Biogeography 20: 79-85. Haase, P. 1994. Genetic relationships and inferredevolutionarydivergenceinthe New Zealand taxa of Nothofagus. Results from isozyme analysis. Australian Systematic Botany 7:47-55. Hasbún R, Iturra C, Moraga P, Wachtendorff P, Quiroga P, Valenzuela S. 2011. An efficient and reproducible protocol for production of AFLP markers in tree genomes using fluorescent capillary detection.Tree Genetics and Genomes. DOI: 10.1007/s11295-011-0463-6. Hinojosa L. 2005. Cambios climáticos y vegetacionales inferidos a partir de paleofloras cenozoicas del sur de Sudamérica. Revista geológica de Chile 32(1):95-115. INFOR. 2011. Situación actual del sistema de abastecimiento de semillas y producción de plantas nativas en viveros de la zona centro-sur del país. Informe preliminar proyecto 028/2010 “Fundamentos para una normativa sobre origen de semillas y calidad deplantas forestales nativas”. 1er Concurso del Fondo de Investigación del Bosque Nativo. INN,2006. Norma Chilena Oficial. NCh 2957. Of2006.Material de propagación de uso forestal. 1ra ed.Instituto Nacional de Normalización, Santiago. INTA 2012. Artículo de divulgación - porAcuña C,. Azpilicueta M,. Fernandez P,.Gallo L,. Gonzalez S,. Hopp H,. Marchelli P,. Rivarola M,. Pomponio M,.PaniegoN,. Torales S. [en linea]. Publicado originalmente: 14 de agosto de 2012., [fecha de consulta 13 de abril de 2013]. Disponible en: http://inta.gob.ar/proyectos/aebio-242421. IPCC. 2007. Climate Change 2007: Impacts, Adaptation and Vulnerability. Contribution of Working Group II to the Fourth Assessment Report of the Intergovernmental Panel on Climate Change, M.L. Parry, O.F. Canziani, J.P. Palutikof, P.J. van der Linden and C.E. Hanson, Eds, Cambridge University Press, Cambridge, UK, 976pp. IPGRI, FRIM, Kuala Lumpur, Malasia. Jiménez P, Collada C. 2000. Técnicas para la evaluación de la diversidad genética y su uso en los programas de conservación. Investigación Agraria: Sistemas y Recursos Forestales, Fuera de Serie (Chile) 2: 237-248. Jones R., Vaillancourt R., Jordan G. 2004. Microsatellites for use in Nothofagus cunninghamii (Nothofagaceae) and related species.Molecular Ecology 4:1-14. Karp, A. 2000. The new genetic era: will it help us in managing genetic diversity? SAT 21 Meeting. Kleinschmit, J, Kownatzki D, Gregorius H. 2004. Adaptational characteristics of autochthonous populations - consequences for provenance delineation. Forest Ecology and Mangement 197:213-224. Lawler J, White D, Neilson, Blaustein A, Andrew R. 2006. Predicting climate-induced range shifts: model differences and model reliability. Global Change Biology 12(8):1568-1584. Lenoir J, Gégout J, Marquet P,de Ruffray P, Brisse H. 2008. A significant upward shift in plant species optimum elevation during the 20th century. Science 320(5884):1768-1771. Lindgren, D, Ying, C. 2000. A model integrating seed source adaptation and seed use. New Forests 20:87-104. Lomolino M, Riddle B, Brown. 2006. Biogeography. Third Edition. Sinauer. 845 pp. Lu Y, Yan J, Guimaraes CT, Taba S, Hao Z, Gao S, Chen S, Li J, Zhang S, Vivek BS, Magorokosho C, Mugo S, Makumbi D, Parentoni SN, Shah T, Rong T, Crouch JH, Xu Y. 2009. Molecular characterization of global maize breeding germplasm based on genome-wide single nucleotide polymorphisms. Theor Appl Genet 120:93–115. Lu F., Lipka A., Glaubitz J., Elshire R, Cherney J., et al. 2013. Switchgrass Genomic Diversity, Ploidy, an.d Evolution: Novel Insights from a Network-Based SNP Discovery Protocol. PLoS Genet 9(1): e1003215. doi:10.1371/journal.pgen.1003215. Manos P., Nixon K., Doyle. J. 1993. Cladistic analysis of restriction site variation within the chloroplast DNA Inverted repeat region of selected Hamamelididae. Australian SystematicBotany18: 551-562. ManosP., 1997. Systematics of Nothofagus (Nothofagaceae)basedon rDNA spacer sequences (ITS): taxonomic congruence with morphology and plastid sequences. American Journal of Botany84:1137. Manos, P.S., Steele. K, 1997. Phylogenetic analyses of “higher” Hamamelididae based on plastid sequence data. American Journal of Botany 84: 1407-1419. Marchelli P., Gallo L., Scholz F., Ziegenhage B. 1998. Chloroplast DNA markers reveal a geographical divide across Argentinean southern beech Nothofagus nervosa (Phil.) Dim. et Mil. distribution area. Theoretical and Applied Genetics97: 642—646. Marchelli P., Gallo L.2000a. Genetic analysis of isozyme variants in open pollinated families of Southern beech Nothofagus nervosa (Phil.) Dim.et Mil. Silvae Genetica 49: 90-98. Marchelli P., Gallo L. 2000b. Variación aloenzimatica, de ADN de cloroplasto y de ADN nuclear en poblaciones y progenies de Raulí en argentina. Revista Domesticación ymejoragenéticaenRaulíyRoble. Editorial Maval Limitada. ISBN: 956 288-691•3 Registro de Propiedad Inlelectual N°115.794 Agosto 2000.p 157-180. Marchelli, P., Gallo, L. 2001. Genetic diversity and differentiation in a southern beech subjected to introgressive hybridisation. Heredity 87: 284-293. Marchelli,P. 2002. Variabilidad genética en Raulí (Nothofagus nervosa (Phil.) Dim etMil.),su relación con procesos evolutivos y la importancia en la conservación y utilización de sus recursos genéticos. Tesis Doctoral, Centro Regional Universitario Bariloche, Universidad Nacional del Comahue. 222 p. Marchelli, P., Gallo, L. 2004. Conservación y utilización de la variación genética en poblaciones argentinas de Raulí (Nothofagus nervosa (Phil.) Dim. et Mil.).IUFRO International Symposium, Valdivia, Chile.Publicación en CD. 17 p. Marchelli, P., Gallo, L.2004b. The combined role of glaciation and hybridization in shaping the distribution of genetic variation in a Patagonian southern beec.Journal of Biogeography Volume 3: 451–460. Marchelli P., Gallo L. 2006. Multiple ice-age refugia in a southern beech of South America as evidenced by chloroplastDNA markers. Conservation Genetics 7:591-60. Marchelli,P., Caron,H., Azpilicueta,M and Gallo, L. 2007. A New Set of Highly Polymorphic Nuclear Microsatellite Markers for Nothofagus nervosa and Related South American Species. Mathiasen P, AC Premoli. 2010. Out in the cold: genetic variation of Nothofagus pumilio (Nothofagaceae) provides evidence for latitudinally distinct evolutionary histories in austral South America. Molecular Ecology 19:371-385. Martin P., Dowd J. 1993. Using sequences of rbcL to study phylogeny and biogeography of Nothofagus species. Australian Systematic Botany 6: 441-447. Martín M. ,Muñoz S., Muñoz F., Uribe M., Molina J., Herrera M., Martína L., Álvarez J. 2010. Primeros resultados en el desarrollo de un marcador genético basadoen las proteínas de reserva en dos especies del género Nothofagus. Bosque 31: 252-257. Martinez G., Zappacosta D., Arena M., Curvetto N. 2001. Changes in isoperoxidase patterns during the invitro rooting of Nothofagus antarctica. Plant Physiology 27: 43–53. Mattioni C., Casasoli M., González M., Ipinza R., Villani F.2002. Comparison of ISSR and RAPD markers to characterize three Chilean Nothofagus species. Genetic 104: 1064-1070. Mesén F, Guevara AL, Jiménez ML. 1996. Guía técnica para la producción de semilla forestal certificada y autorizada. Serie Técnica, Manual Técnico No. 20. CATIE-Oficina Nacional de Semillas-MINAE. Turrialba, Costa Rica. 34 pp Nimmakayala P., Levi A., Abburi L., Lakshmi V., Abburi L., Tomason Y.,Saminathan T.,Gopinath V., Malkaram S.,Reddy R.,Wehner T., Mitchell S., Reddy U. 2014. Single nucleotide polymorphisms generated by genotyping by sequencing to characterize genome-wide diversity, linkage disequilibrium, and selective sweeps in cultivated watermelon. BMC Genomics 15:767. Nuroniah HS. 2009. Diagnostic markers for the identification of the tree species Shorea leprosula Miq. And S. parvifolia Dyer and the geographic origin of S. leprosula Miq. [Dissertation]. Goettingen University, Goettingen. Alemania. OCDE. 2007. OECD Scheme for the Certification of Forest Reproductive Material moving in International Trade. OCDE, Paris. 26 pp. Pastorino M.,Marchelli P., Milleron M., Gallo L. 2008. Inheritance of isozyme variants in Nothofagus antarctica (G.forster) Oersted. Journal of Basic and Applied Genetics 19: 27-33. Pastorino M., Marchelli P., Milleron M., Soliani C., Gallo L. 2009.The effectof different glaciation patterns over the current genetic structure of the southern beech Nothofagus antarctica.Genética 136:79-88. Petit J, Raynaud D, Basile I, Chappellaz J, Davisk M, Ritz C, Delmotte M, Legrand M, Lorius C, Pe L, Saltzmank E. 1999. Climate and atmospheric history of the past 420000 years from the Vostok ice core , Antarctica. Nature 399:429-435. Pineda, G. 2000a. Variabilidad aloenzimatica de N. alpina en chile.Revista Domesticación y mejora genética en Rauli y Roble. Editorial Maval Limitada. ISBN:956 288-691-3 Registro de Propiedad Inlelectual N° 115.794 Agosto 2000. Páginas 95-120. Pineda, G. 2000b.Variabilidad isoenzimatica del huerto semillero clonal de N. alpina. Editorial Maval Limitada. ISBN: 956288-691-3 Registro de Propiedad Inlelectual N" 115.794 Agosto 2000. Páginas 297 -306. Premoli, A. 1996.Allozyme polymorphisms, ourcrossing rates, and hybridization of South American Nothofagus. Genética 97: 55-64. Premoli, A. 1997. Genetic variation in a geographically restricted and two widespread species of South American Nothofagus. Journal of Biogeography24:883–892. Premoli A. 1998. The use of genetic markers to conserve endangered species and to design protected areas of more widespread species. In Proceedings of an International Workshop on Recent Advances in Biotechnology for Tree Conservation and Management (International Foundation for Science, ed.). Santa Catarina, Brazil, Universidade Federal de Santa Catarina. p. 157-171. Premoli, A. 2003. Isozyme Polymorphisms Provide Evidence of Clinal Variation With Elevation in Nothofagus pumillo. Journal of Heredity 94:218–226. Premoli, A and Kitzberger, T. 2005. Regeneration mode affects spatial genetic structure of Nothofagus dombeyi forests. Molecular Ecology 14:2319–2329. Schultz L. 2008. Conservation Genetics of a Gondwana relict rainforesttree, Nothofagus moorei (F.Muell)Krasser. Tesis Doctoral. Universidad de la Tecnología de Queensland. Australia. Searle R, Rovira R. 2008. Cambio climático y efectos en la Biodiversidad: el caso chileno. En: CONAMA. 2008. Biodiversidad de Chile. Patrimonio y Desafíos, pp 502-505. Setoguchi, H.,Ono, J., Doi, Y., Koyama, H.,Tsuda, M. 1997. Molecular phylogeny of Nothofagus (Nothofagaceae) based on the atpB-rbcL intergenic spacer of the chloroplast DNA. Journal of Plant Research 110: 469-484. Soliani C., Sebastiani F., Marchelli P., Gallo L., Vendramin G. 2010. Development of novel genomic microsatellite markers in the southern beech Nothofagus pumilio (Poepp. et Endl.) Krasse. Molecular Ecology Resources 10:404-408. Soto DP, Ríos AI. 2009. Seis años de respuesta de una plantación intensiva de Raulí y Coihue en la Precordillera de la Costa. Revista Bosque Nativo 44: 7-11. Stecconi M.,Marchell iP.,Puntieri J., Gallo L. 2002. Hybridisation between Nothofagus antarctica (deciduous) and N. dombeyi (evergreen) (Nothofagaceae) in natural communities. In Southern Connection Bulletin 21: 8-9. Steconni M.,Marchelli P., Puntieri J.,Picca P., Gallo L. 2004. Natural hybridisation between a deciduous (Nothofagus antarctica,Nothofagaceae) and an evergeen (N. dombeyi) forest tree species as evidenced by morphological and isoenzymatic traits. Annals of Botany 94:775-786. Stöckler K. 2001. Origins and Evolution of the New Zealand Forest flora a molecular phylogenetic approach. Tesis Doctoral. Universidad de Massey. New Zealand. Torales S.,Rivarola M., Pomponio M., Fernandez P., Acuña C., Marchelli P., Gonzalez S., Azpilicueta M., Hopp H., Gallo L., Paniego N., Marcucci S. 2012. Trascriptome survey of Patagonian southern beech Nothofagus nervosa (=N. alpina): assembly,annotationand molecular markers discovery. BMC Genomics 13: 291. Thomsen T. 2002. Biogeography of Nothofagus subgenus Fuscospora in the South Island of New Zealand inferred from chloroplast DNA. Tesis Doctoral. UniversidaddeCanterbury. Nueva Zelanda. Villagrán C, Hinojosa L. 1997. Historia de los bosques del sur de Sudamérica, II: Análisis fitogeográfico. Revista Chilena de Historia Natural 70: 241-267. Vergara R., Gitzendanner M., Soltis D., Soltis P. Population genetic structure, genetic diversity, and natural history of the South American species of Nothofagus subgenus Lophozonia (Nothofagaceae) inferred from nuclear microsatellite data. John Wiley & Sons Ltd. Walther G, Post E, Convey P, Menzel A, Parmesan C, Beebee T, Fromentin J, Hoegh-Guldberg O, Bairlein F. 2002. Ecological responses to recent climate change. Nature 416:389-395. Woodward F. 1987.Climate & Plant Distribution.Cambridge Studies in Ecology.Cambridge University Press. UK. 174 p. Zobel B, Talbert J. 1984. Applied Forest Tree Improvement. John Wiley and Sons, IAC. USA. |
068/2012 Proyecto Certificación Genética del Origen de Materiales Reproductivos de Coigüe mediante Herramientas Holeculares y Modelos Ecológicos |
Financiado por el Fondo de Investigación del Bosque Nativo de la Corporación Nacional Forestal - Desarrollado por la Universidad de Concepción |
Contacto: e-mail origen_coigue@udec.cl - Fono +56-41-2204991 |