Las Moléculas Grandes
Breve descripción de las Macromoléculas Biológicas
Las macromoléculas biológicas son un pre requisito para todas las formas de vida. Sus pesos moleculares oscilan entre 104 y 106, y están formadas principalmente por C, N, O, H, P y S. Sólo un miembro de este grupo de macromoléculas, y más precisamente, el ADN, por su estructura ordenada, puede ser capaz de conducir la información genética que determina a su vez, la secuencia de ámino ácidos de las proteínas y por lo tanto las características específicas de cada especie. La secuencia de las subunidades componentes, (Monómeros), organizadas en estas moléculas grandes, es responsable de la transmisión de la información genética, de las funciones de las células, proporcionan además los mecanismos de regulación de la estructura espacial en condiciones naturales, es decir, en un rango de concentraciones de pH de 6.5-7.7, temperaturas entre 10 y 40ºC, concentraciones iónicas débiles, 760 mbares de presión, con las excepciones correspondientes. La función de estas macromoléculas es claramente dependiente de y determinada por la formación de una única y correcta organización espacial. Por esta razón, resulta imprescindible en la actualidad, conocer la estructura de estas biomoléculas, las fluctuaciones que les son permitidas para realizar su función y la fuerte dependencia de estas fluctuaciones con los factores ambientales, es decir, su comportamiento tanto en solución, como en interacción estrecha con otras moléculas de los sistemas vivientes como de su entorno.
La síntesis de estas macromoléculas en los sistemas biológicos corresponde a la condensación de subunidades (monómeros), de pesos moleculares pequeños. Pueden reconocerse dos clases principales, dependiendo de sus características globales:
2.1. Macromoléculas formadas por un gran número de monómeros idénticos unidos linealmente, como amilosa y celulosa, o en estructuras ramificadas como el glicógeno. Los más comunes son los polisacáridos, en que la unidad monomérica típica es

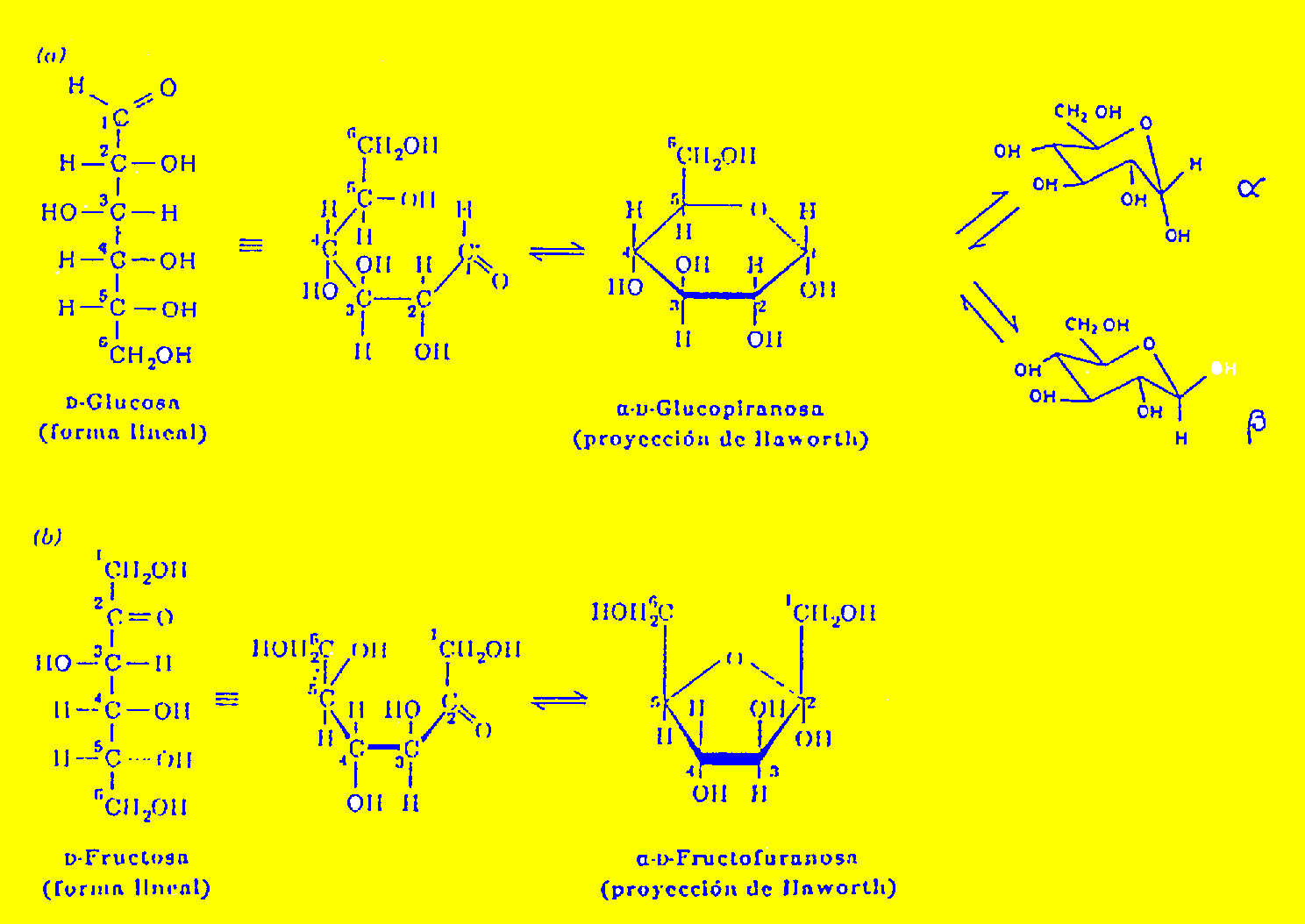
Esquema 2.1. a) Configuración y sistemas de nomenclatura utilizados. Ejemplos: Glucosa y Fructosa.Si una cadena de azúcar es suficientemente larga, uno de los grupos OH se adiciona al grupo carbonilo para producir un hemiacetal cíclico.
b) Distinción entre y -glucopiranosa.
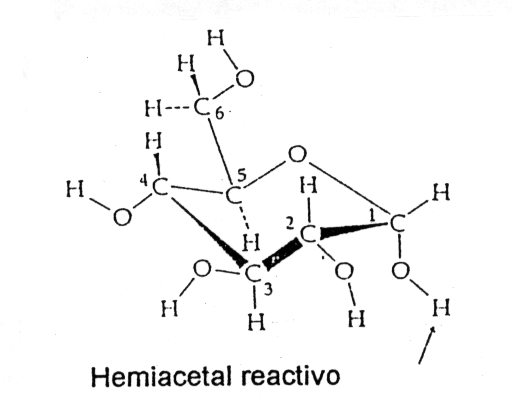
Figura 2.1. Estructura de a-D-glucosa.
A pesar de la gran variedad de unidades monoméricas que existen en las cadenas de carbohidratos (ver Esquema 2.1 y Esquema 2.2), las posibilidades conformacionales son pocas. En los polisacáridos las uniones entre azúcares en general ocurre entre el C1 de un anillo y el C4 o C6 del siguiente. El anillo del azúcar es una estructura relativamente rígida y la unión entre dos de estas subunidades puede ser identificada principalmente a través de sus ángulos de torsión f y j. Por convención se usa f = j = 0, cuando los planos que pasan por el centro del anillo son coplanares. Esto puede observarse en la Figura 2.2. El símbolo -1, 4 indica que este disacárido conecta el Carbono 1 de un anillo con el Carbono 4 del otro, y que la configuración alrededor del Carbono anomérico es a.
Figura 2.2. Dos moléculas de -1, 4 D-glucosa unidas por un enlace glicosídico.
Esquema 2.2. Estructura química de los monosacáridos más comunes. Los azúcares contienen varios centros quirales y a los diasteroisómeros se les asigna nombres distintos. Observar por ejemplo glucosa, manosa y galactosa. Cada uno de los azúcares existe como un par de enantiómeros D y L, imágenes especulares uno del otro.
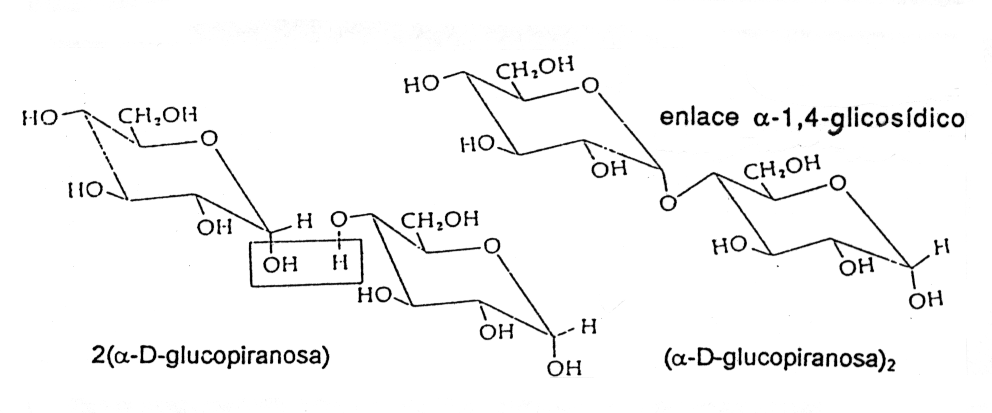 Figura 2.3. Formación de un disacárido a partir de subunidades monoméricas.
Figura 2.3. Formación de un disacárido a partir de subunidades monoméricas.
La Figura 2.3. muestra la reacción de formación de un disacárido, maltosa, a partir de dos subunidades de glucosa. Una adición similar de otras unidades de glucosa conduciría a la formación de un polímero. Los grupos funcionales presentes en los azúcares, en su mayoría son -OH, es decir que las posibilidades de variación en los polisacáridos es bastante alta, ya que dependiendo del azúcar, existirán 4, 5 o 6 grupos hidroxilos por unidad monomérica sin embargo las uniones que se encuentran en los polisacáridos son generalmente del tipo a-1,4 a-1, 6 o b-1, 4 glicosídicas. Esto permite la existencia de polisacáridos lineales y ramificados. Sus características en solución serán diferentes dependiendo de si éstos son lineales o no, del tipo de subunidades, del peso molecular (o número de subunidades monoméricas n), y del tipo de modificaciones presentes en ellos (-COOH, -NH2, -SO4, -NHCOCH3).
El tipo de estructura de estos polisacáridos está estrechamente relacionada con la función específica que estos compuestos tengan, ya que de su estructura dependen sus propiedades de solubilidad. Entre los polisacáridos lineales encontramos a la amilosa, un componente de almidón de las plantas formado por unidades de a-D-glucopiranosa enlazadas por uniones 1,4. En general, mientras más grande sea la macromolécula y más ramificada, disminuyen sus posibilidades de ser solubles en medio acuoso. Esto resulta particularmente importante si pensamos que la celulosa y la quitina proporcionan sostén a las plantas e insectos respectivamente. La celulosa es una poliglucosa b-1, 4; la quitina es un polímero lineal b 1,4 de N-acetil-glucosamina, otros en cambio, como amilopectina, glicógeno que a pesar de estar altamente ramificados son solubles.
El glicógeno es un polímero ramificado que está formado por un esqueleto lineal a-1,4 de a-D-glucopiranosa, con ramificaciones a través de enlaces a-1,6. Las posiciones de unión y ramificaciones de algunos de estos compuestos se muestran en la Figura 2.4:
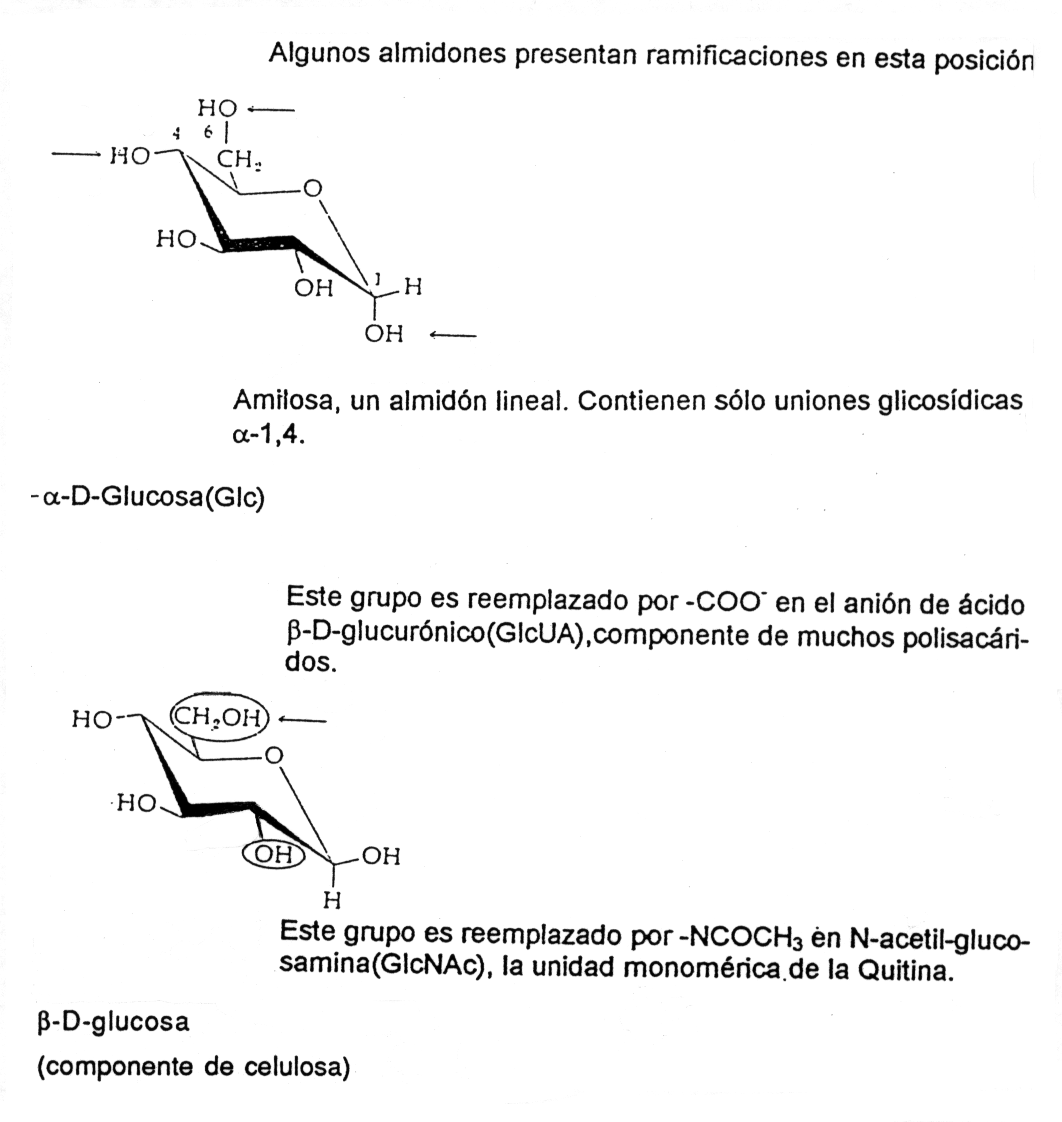
Figura 2.4. Estructura de algunas unidades de monosacáridos indicando las posiciones en que pueden ocurrir ramificaciones.
Varias de estas macromoléculas, por su función y por su estructura, como ya se ha señalado, se no son particularmente solubles ya que requieren constituir sostén para otras funciones; otras que constituyen almacenamiento de unidades de a-D-glucosa, para ser utilizada en los procesos metabólicos celulares se encuentran altamente hidratadas, aunque su movilidad se encuentra restringida debido al tamaño de la molécula. Por lo tanto generalmente se encontrarán localizadas. Los grupos que se encuentran en la superficie interaccionarán con el solvente formando una capa de agua organizada a su alrededor, e interaccionará con otras macromoléculas, metabolitos pequeños y iones presentes en el medio, dependiendo del tipo de subunidades que lo compongan.
Anexo 2.1.
Tipos de azúcares y de uniones en polisacáridos
Homopolisacáridos
Celulosa : poliglucosa b-1,4
Quitina : Polímero lineal b-1,4 de N-acetil-glucosamina
Pectina : Poli b-1,4 ácido carboximetil-galacturónico
Almidón : Polisacárido de reserva en plantas, contiene amilosa y amilopectina
Amilosa : Polímero lineal de a-D-glucopiranosa
Glicógeno : Polisacárido de reserva en animales. Formado por una cadena lineal de a-1,4 D-glucosa con ramificaciones a-1,6.
Dextrano : Poli D-glucosa a-1,6 con ramificaciones a-1,4.
Heteropolisacáridos
Constituidos por más de un tipo de monosacáridos, por ejemplo aquellos constituidos por dos monosacáridos en secuencia alternante
Acido hialurónico : componente del cemento celular en el tejido conectivo.
(-bGlcUA-(1,3)-bGlcNAc-(1,4))n
(Acido D-glucurónico-N-acetil-D-glucosamina)
Condroitin sulfato: (-bGlcUA-(1,3)-bGalNAc-(1,4))n
(Acido D-glucurónico-N-acetil-galactosamina)
Muchos de los carbohidratos se encuentran asociados a proteínas para efectuar su función, así podemos encontrarlos en las secreciones celulares como el mucopolisacárido que contiene una mezcla de carbohidratos como el condroitin sulfato o la Heparina que posee propiedades anticoagulantes y que contiene unidades disacáridas [(-ácido urónico(1-4)-GlcN-2, 6 disulfato-(1-4)]n.
Otros polisacáridos
a) Residuos Galacturonosil unidos a través de enlaces a-D-1,4 como los encontrados en pectina
Algunos de los grupos carboxilos se encuentran esterificados. Muchas veces es posible encontrar residuos de ramnosa y/o arabinosa intercalados.
b) Estructura parcial de un arabinoxilano de angiospermas
c) Elementos estructurales de una parte de una molécula de guaiacil lignina
R representa el resto de la molécula de lignina. Aunque no se sabe completamente la estructura de la lignina si este esquema permite representar el tipo de uniones que se encuentran en ella.
Los carbohidratos también se encuentran formando parte de glicoproteínas, de membranas proporcionando a las proteínas una aun mayor diversidad a causa de los monómeros particulares, en general a través de enlaces del tipo O-glicosídicos unidos a través de residuos de treonina y serina o por uniones N-glicosídicas a través de asparragina y glutamina. Los carbohidratos pueden considerarse como monómeros polares y por lo tanto se encuentran generalmente expuestos al solvente(agua) e interaccionando con él.
2.2. Macromoléculas formadas por un número predeterminado de diferentes monómeros, ubicados en un arreglo lineal ordenado. Proteínas y Acidos Nucleicos
2.2.1. Acidos Nucleicos
Los ácidos nucleicos, ADN y ARN son polinucleótidos. La estructura básica de repetición son los nucleótidos. Cada nucleótido está formado por una base púrica o pirimidínica, un azúcar(D-ribosa o D-2-desoxiribosa) y por el ácido fosfórico. Esta estructura está representada en la Figura 2.5.
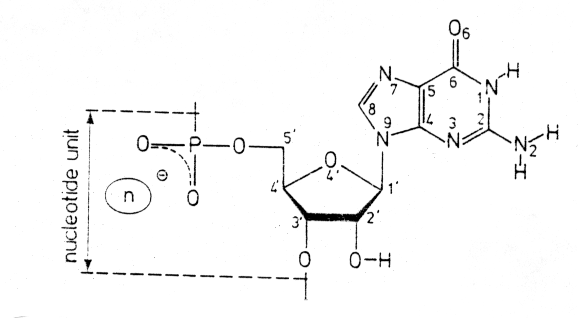
Figura 2.5. Estructura de un nucleótido. Los átomos están numerados del 1 al 9 en la base y del 1' al 5' en el azúcar.
Los nucleótidos se unen por eliminación de una molécula de agua como se indica en la figura 2.6.
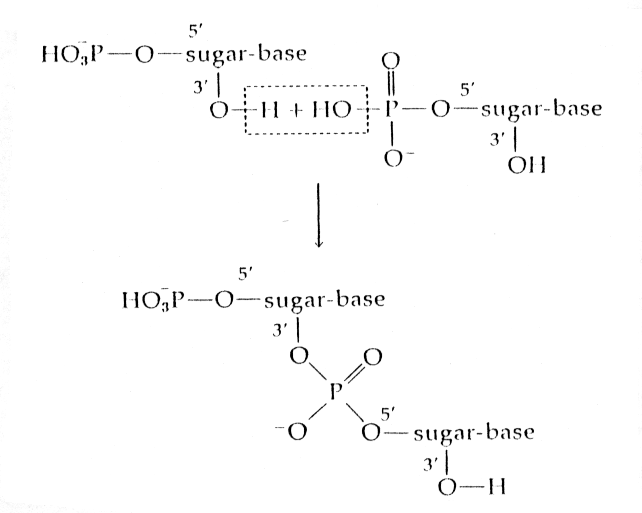
Figura 2.6. Reacción de formación de un dinucleótido. Para la formación de un polinucleótido se repite la misma reacción con eliminación de una molécula de agua por nucleótido adicionado.La unión entre nucleótidos se produce por enlaces fosfodiester entre el 5'OH de un azúcar y el 3'OH del siguiente.
A los heterociclos purinas y pirimidinas que se encuentran en los ácidos nucleicos, se les conoce en general como bases (ver Figura 2.7), aunque no son específicamente básicos. Los nucleósidos son los N-glicosil derivados de las bases con D-ribosa o D-2-desoxiribosa. Los nucleótidos son los ésteres fosfato de los nucleósidos.
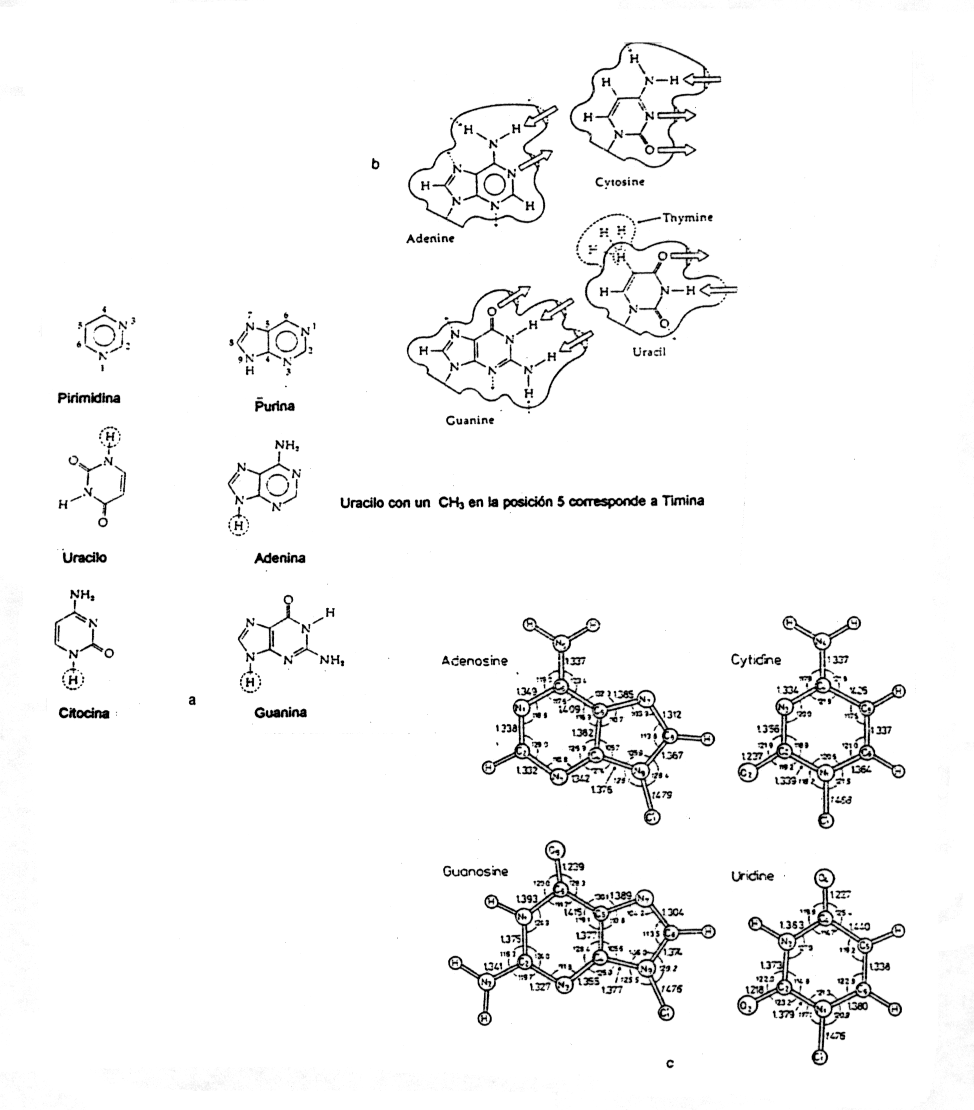
Figura 2.7.
a) Estructura esquemática de las bases nitrogenadas que se encuentran en los ácidos nucleicos, el sitio de unión de la base al azúcar está indicado con un círculo sobre el H.
b) Los sitios de posible formación de los puentes hidrógeno se indican con una flecha. La dirección de la flecha indica el aceptor.
c) Longitudes y ángulos de enlace de las bases nucleotídicas.
Tomado de Metzler, D. Biochemistry. (1977) Academic Press, New York y de Saenger, W. Principles of Nucleic Acid Structure (1988) Springer Verlag, Berlín.
De acuerdo a lo destacado en la figura, las bases nitrogenadas son planares y pueden encontrarse como keto o ámino tautómeros. Los grupos ámino son coplanares con los héterociclos debido a la resonancia electrónica.
Figura 2.8. Estructura de ribosa y desoxiribosa.
El anillo furanosa de la ribosa y de la desoxiribosa no son planares y pueden existir en diferentes conformaciones. Generalmente es el C-2' o el C-3' el que está fuera del plano de los otros cuatro átomos del anillo. Si este carbono está por encima del anillo se denomina endo; si está por debajo se denomina exo. La conformación C-2' endo y la C-3' endo parecen ser las más comunes en los nucleótidos sin polimerizar. En cualquiera de los dos casos puede encontrarse las conformaciones tipo N (north) o S (south) que corresponden al esquema que se muestra en la Figura 2.9
Figura 2.9. Representación esquemática de los tipos más comunes de conformaciones encontradas en los azúcares de los nucleótidos. La nomenclatura S y N fue asignada por los espectroscopistas. Se ha determinado que puede haber transiciones rápidas N´S a través de un movimiento vertical.
Las conformaciones más comunes
para una hexosa como la glucosa
se esquematizan en el esquema
adjunto y se muestran además
los equilibrios correspondientes.
Figura 2.10. Estructura de un nucleótido.
Se define la conformación anti (127º), en que el CO y el NH en las posiciones 2 y 3 del anillo pirimidínico (o en las posiciones 1, 2 y 6 del anillo de purina) se alejan del azúcar, y la conformación syn, (con una diferencia de 180º), en que estos grupos se encuentran sobre el anillo. En general, para describir con detalle la conformación de un polinucleótido se requiere conocer otros ángulos que relacionan las diferentes partes de la molécula.
Son las características de las subunidades monoméricas, en que algunos de sus componentes son planares y que presentan grupos cargados negativamente como el fosfato, las que permiten al polímero adoptar varios tipos de conformaciones, así como interaccionar con otras moléculas del medio.
Una cadena polinucleotídica puede representarse abreviadamente por convención de la siguiente manera:
5'G G C U U U A C3'
5'GpGpCpUpUpUpApC3'
que corresponde a los nucleótidos Guanosina, Guanosina, Citosina, tres nucleótidos de Uracilo, Adenosina y Citosina, escritos desde el extremo 5' hasta el 3'. Esto último indica la numeración correspondiente a los átomos de carbono en la ribosa.
En la investigación sobre la estructura de los ácidos nucleicos, se han utilizado técnicas como la cristalografía por difracción de rayos X. El método convencional de cristal único se utiliza para el estudio de mono y oligo nucleótidos. Para el t-ARN (ARN de transferencia), que tiene una estructura globular, se utiliza los métodos aplicados a la cristalografía de proteínas. ADN y ARN polimérico no forman cristales, sólo pueden obtenerse fibras cuasicristalinas debido al desorden propio de las estructuras fibrilares, y por lo tanto la información que puede obtenerse de la cristalografía es mucho menor. Así, ha sido posible proponer modelos estructurales basados en el conocimiento estereoquímico de las unidades pequeñas utilizando además, cálculos de energía potencial. Entre los métodos espectroscópicos, la Resonancia Magnética Nuclear es la metodología que ha aportado una mayor cantidad de información sobre la conformación de mono y oligonucleótidos en solución, Estos datos combinados con la información cristalográfica y los cálculos teóricos producen un modelo coherente con los datos experimentales.
Acido desoxiribonucleico (ADN)
El ADN es una molécula extremadamente larga y delgada. La hebra de ADN que constituye el genoma del Escherichia coli tiene 1.1 mm de longitud y corresponde a un peso molecular de 2.8x109. Está compuesto de nucleótidos enlazados por uniones fosfodiester, tal como lo hemos descrito. Estos nucleótidos contienen 4 distintos tipos de bases; estas son Guanina (G), Citosina (C), Adenina (A) y Timina (T). El modelo que hoy tenemos del ADN, está basado en el descubrimiento de Watson y Crick que se publicó en 1953, en el que proponen al ADN como una doble hélice de dos cadenas polinucleotídicas antiparalelas y complementarias, en que el apareamiento de las bases se realizaba a través de uniones puente H. Existen dos modelos de apareamiento de las bases nucleotídicas (Watson y Crick y el modelo de Hoogsteen). En la Figura 2.11 se muestra el modelo de Watson y Crick para el apareamiento de las bases indicando sus dimensiones. En esta figura es posible observar que las dimensiones de las uniones puente hidrógeno no son idénticas, indicando la "flexibilidad" del enlace y del efecto que las propiedades de la vecindad atómica tiene sobre la fuerza de este tipo de interacciones:
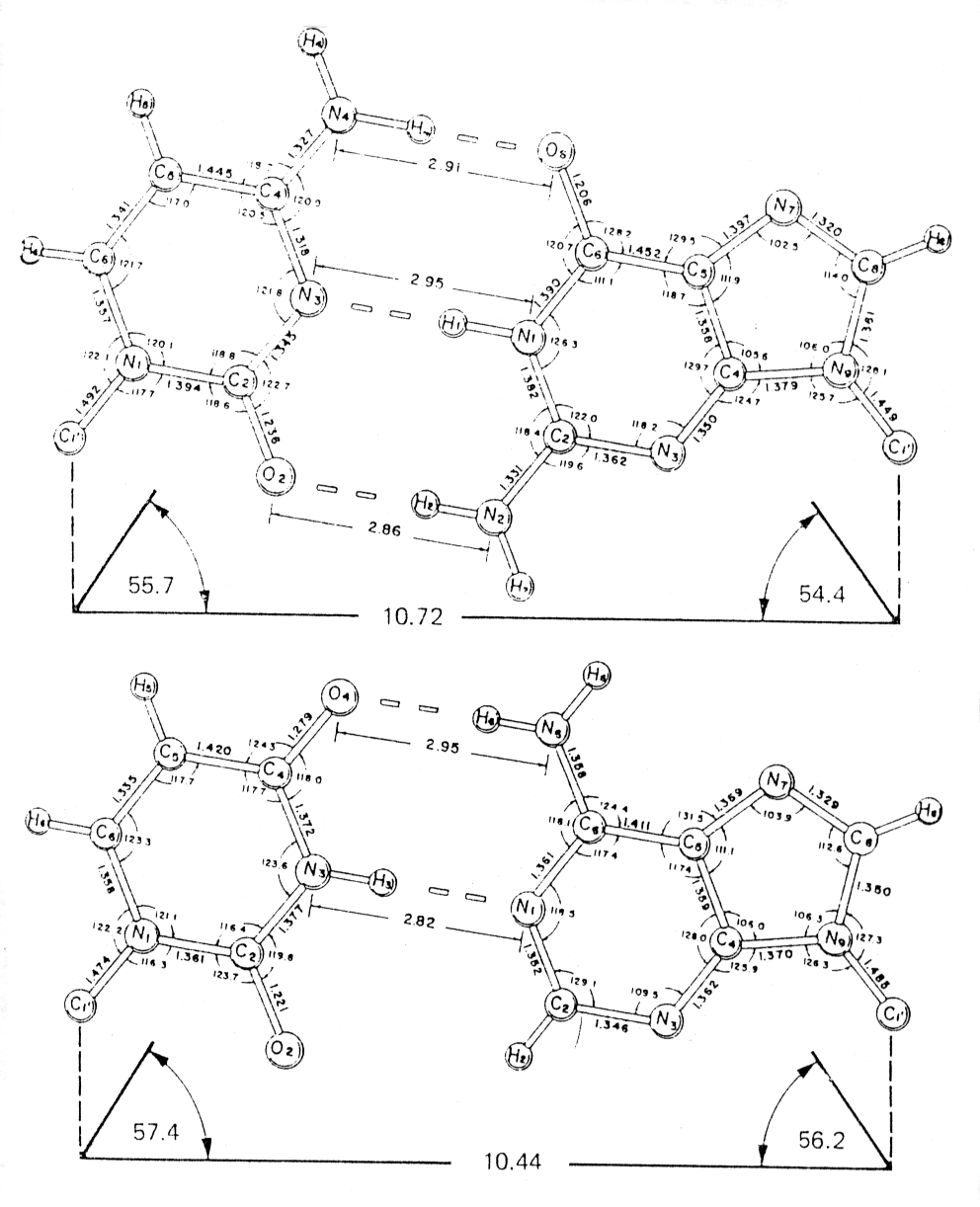
Figura 2.11. Modelo de Watson y Crick para el apareamiento de las bases nucleotídicas. Arriba se muestra el par GpC, abajo el par ApU cuyas dimensiones han sido determinadas por difracción de rayos X.Tomado de Saenger, W. Principles of Nucleic Acid Structure(1988) Springer Verlag, Berlín.
Figura 2.12. Vista aplanada de modelo de Watson y Crick para la estructura de un fragmento de ADN.
Anexo 2.2.
Los sistemas heterocíclicos en los nucleósidos son susceptibles de sufrir tautomerismo; dependiendo de cuál sea el tautómero presente, así serán el tipo y número de uniones puente hidrógeno que podrán formarse. Esto se indica en detalle en la figura que indica el tautomerismo Ceto-Enol y Amino-Imino en bases nucleotídicas. Puede observarse que en la forma enólica, G se hace equivalente a A y U equivalente a C. En la forma imino A es equivalente a U o G y C es equivalente a A. En condiciones fisiológicas sin embargo, más del 99.99% corresponde a la forma Ceto o amino, lo cual favorece la función del ADN.
La protonación o deprotonación de las bases ocurre entre pH 3.5 y 10.5; esto hace que el pH sea un componente decisivo en el tautomerismo de estas bases. Algunos valores de pKa para las bases nucleotídicas se muestran en la siguiente tabla:
Tabla de pK
|
Compuesto |
Nucleósido |
3'P |
5'P |
|
|
Adenosina/N1 |
3.52 |
3.70 |
3.88 |
|
|
Citidina/N3 |
9.38 |
9.96 |
10.06 |
|
|
Guanosina/N1 Uridina/N3 |
9.42 4.17
|
9.84 4.13
|
10.00 4.54 |
Este tipo de apareamiento ocurre solamente si A se aparea con T y C con G(ver figura 2.12). De este modo una de las cadenas es complementaria pero no idéntica a la otra. La secuencia de bases en el ADN deriva de un patrón genético para cada especie. El ADN puede adoptar varias conformaciones dependiendo de las condiciones ambientales como presencia de contraiones, humedad relativa, secuencia y composición nucleotídica. La Figura 2.13 muestra el modelo más difundido de la doble hélice del ADN, el B-DNA, indicándose la nomenclatura usual para denominar diferentes aspectos de la molécula.
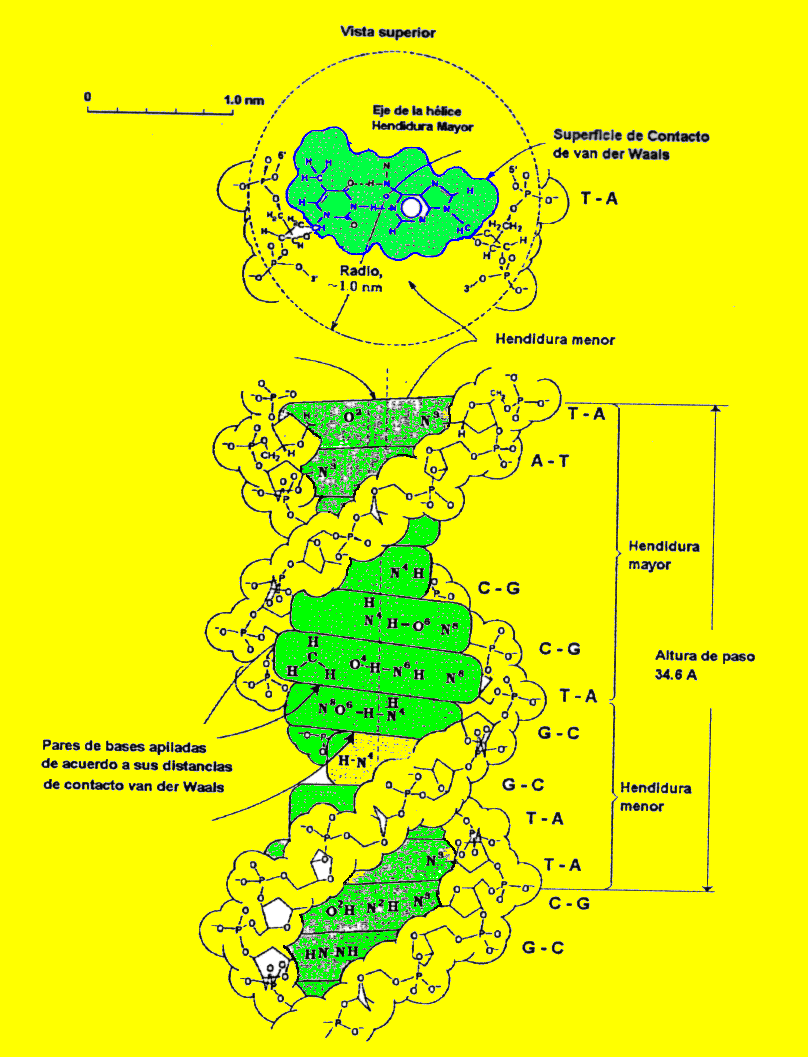
Figura 2.13. La doble hélice de ADN (forma B).
Tomado de Arnott, S. a Hukins, D.W.L.,
J.Mol. Biol. 81: 93-105 (1975).
El modelo establece que se trata de una doble hélice antiparalela hacia la derecha. Presenta dos curvaturas, denominadas hendidura mayor y hendidura menor de acuerdo a su tamaño. Los grupos fosfato quedan hacia la periferia por lo tanto son susceptibles a la interacción con iones. Cuando la doble hélice está formada su estabilidad es muy alta y son necesarios grandes cambios en el solvente para producir alteraciones en su estructura, durante su formación en cambio la presencia del solvente resulta muy importante. La superficie que expone el ADN resulta fundamental en la interacción con otras moléculas ya que le permite ejecutar su función.
De acuerdo a los modelos propuestos en base a los estudios de difracción de rayos X de fibras de ADN se pueden diferenciar doble hélices a la derecha : A-ADN, B-ADN y T-ADN; y Z-ADN que es una doble hélice a la izquierda. Las características estructurales de cada uno de estos modelos aparecen descritas en Tabla 2.1 y en la Figura 2.14.
Tabla 2.1.
Características Estructurales ideales de A, B, y Z ADN
|
Propiedad |
A |
B |
Z
|
|
Sentido de la hélice |
Arrollada hacia la derecha |
Arrollada hacia la derecha |
Arrollada hacia la izquierda |
|
Diámetro |
26 A |
20 A |
18 A |
|
Pares de bases por vuelta de hélice |
11 |
10 |
12(6dímeros) |
|
Giro de la hélice por par de bases |
33º |
36º |
60º(por dímero) |
|
Elevación por vuelta |
28 A |
34 A |
45 A |
|
Elevación de la hélice por par de bases |
2.8 A |
3.4 A |
3.7 A |
|
Inclinación de bases normal al plano de la hélice |
20º |
6º |
7º |
|
Surco mayor |
Estrecho y profundo |
Ancho y profundo |
Plano |
|
Surco menor |
Ancho y poco profundo |
Estrecho y profundo |
Estrecho y profundo |
|
Atomo desplazado en el azucar Enlace glucosídico |
C(3')-endo
Anti |
C(2')-endo
Anti |
C(2')-endo para pirimidinas y C(3')-endo para purinas Anti para Pirimidinas y syn para purinas |
-------------------------------------------------------------------------------------------------------------
Figura 2.14. Modelo de
a) A-ADN,
b) B-ADN,
c) T-ADN y
d) Z-ADN.
Tomado de Saenger, W. Principles of Nucleic Acid Structure
(1988) Springer Verlag, Berlín.
Los factores que confieren estabilidad y funcionalidad a la molécula de ADN pueden resumirse en los siguientes aspectos:
a) Las interacciones puente hidrógeno entre las bases nucleotídicas permite mantener las dos hebras antiparalelas de ADN unidas, secuestrando las regiones hidrofóbicas hacia el eje de la hélice y permitiendo un apilamiento de bases que favorece las interacciones de corto alcance.
b) Los grupos fosfatos que confieren la carga negativa al ADN quedan expuestos y disponibles para la interacción con moléculas de agua o con proteínas básicas como protaminas o histonas en la formación de cromatina y con otros factores de regulación.
c) El azúcar desoxiribosa presenta un grupo menos que la ribosa que sea susceptible de oxidación o formación de radicales lo que lo hace más estable que el ARN.
d) El tautomerismo de las bases así como las constantes de disociación tanto de las bases como de los grupos fosfato permiten que el cambio de las condiciones ambientales, se favorezca la duplicación del material genético.
Acido ribonucleico(ARN)
La síntesis de proteínas involucra que se haga una copia del ADN genómico en una cadena polinucleotídica distinta, denominada ARN o ácido ribonucleico. Del mismo modo que el ADN, está compuesto por una cadena lineal de nucleótidos con dos diferencias químicas. El azúcar es una D-ribosa y la base Timina es reemplazada por Uracilo.
Las moléculas de ARN no presentan una composición definida de bases y existen como una hebra simple que puede organizarse en una estructura terciaria definida en algunos casos como veremos.Dependiendo de la función biológica, los ARN pueden presentar estructuras alargadas, doble hélices o pueden ser globulares en que algunas regiones son doble hélices conectadas por regiones lineales. Doble hélices cortas se encuentran en t-ARN, r-ARN, en los genes de cubierta proteica de bacteriófagos y en algunos m-ARN. Doble hélices más desarrolladas se encuentran en RNA de virus y en algunos polinucleótidos sintéticos. En todos los casos puede encontrarse el mismo tipo de apareamiento de bases que el que se ha descrito para el ADN.
La composición global de bases de ARN de orígenes diferentes también puede variar. Algunos ejemplos se dan en la Tabla 2.2:
Tabla 2.2. Composición de bases de RNA de orígenes diferentes
|
Procedencia |
% A |
% U |
% G |
% C |
|
E. coli |
24 |
22 |
32 |
22 |
|
Proteus vulgaris |
26 |
19 |
31 |
24 |
|
Euglena |
22 |
21 |
30 |
27 |
|
Virus de la poliomielitis |
30 |
25 |
25 |
20 |
|
Riñon de rata |
19 |
20 |
30 |
31 |
El ARN contiene toda la información del ADN de origen, así como también puede presentar el mismo apareamiento de bases. Este proceso de copia se denomina transcripción y requiere un dinamismo conformacional dependiente de las interacciones entre estos polinucleótidos con enzimas, proteínas componentes de la cromatina en algunos casos, reguladores, iones, etc. También es necesario que pueda liberarse esta copia luego de haberse formado para que el ADN pueda volver a constituirse en una doble hélice.
Este ARN se denomina mensajero(mARN), de una sola hebra y bastante más corto que el ADN, ya que cada uno sólo lleva la información para una proteína. Ya que es una molécula más pequeña que el ADN resulta mucho más soluble. Esto es necesario ya que requiere difundir hacia los ribosomas donde se realiza el milagro de la síntesis proteica. Como sabemos, los codones en el mARN no reconocen directamente los ámino ácidos; éstos tienen que ser presentados por otro tipo de ARN, el ácido ribonucleico de transferencia (tARN). Estos tARN tienen alrededor de 80 nucleótidos, son diferentes para cada ámino ácido pero presentan rasgos estructurales comunes, que pueden resumirse en la Figura 2.15
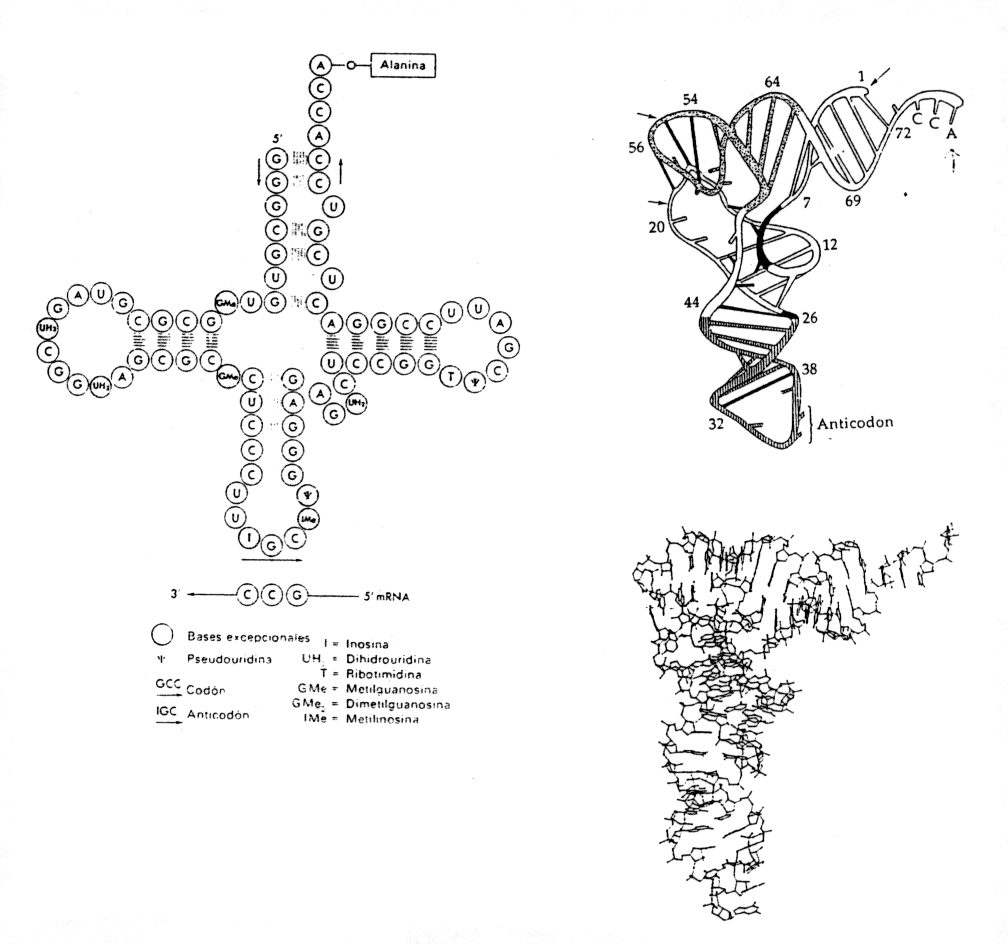
Figura 2.15.
a) Secuencia nucleotídica de alanil-tRNA, que destaca las bases excepcionales y la ubicación del codón-anticodón.b)Plegamiento en L de la secuencia nucleotídica y
c) estructura tridimensional correspondiente al mismo RNA. (Extraído de J.D.Watson en Biología Molecular del Gen, Fondo Educativo Interamericano, 3 edición y de Metzler, D.E. Biochemistry (1977) Acad.Press, N.Y).
El plegamiento que se ha encontrado para la mayoría de los tRNA cuya estructura tridimensional se ha determinado por difracción de rayos X, corresponde lejanamente a una hoja de trébol, en que ciertas regiones han formado puentes hidrógeno, organizándose en estructuras helicoidales, dejando ciertas regiones un poco menos organizadas, regiones de "loops"o bucles sin puentes hidrógeno.
Estas zonas son : El extremo 3', que consta de ACC más un cuarto nucleótido variable; esta es la región a la cual se unen los ámino ácidos. Como puede observarse también existen tres asas. El asa marcada con una flecha contiene el anticodón.
Existen varias otras formas de ácidos ribonucleicos, entre ellos los ARN ribosomales (rARN), es decir ácidos ribonucleicos que se encuentran formando parte del ribosoma. El ribosoma se ha descrito como formado por dos subunidades, clasificadas así de acuerdo a su coeficiente de sedimentación, como la subunidad pequeña(conocida como 16sARN en procariotes y 18sARN en eucariotes), que contiene una sola molécula de rARN de 1600 a 1800 nucleótidos. La subunidad grande (23sARN) contiene alrededor de 2000 nucleótidos y ademas algunos fragmentos pequeños de alrededor de 120 y 160 nucleótidos (5s y 5.8s respectivamente). Esta organización es dependiente de la especie.
Las estructuras de estos ARN, parecen ser bastante conservadas, especialmente las interacciones que estabilizan los bucles, y vueltas. Se ha encontrado que se repiten las secuencias GNRA, UNCG, y CUUG (N = cualquier base) en los bucles formados por 4 nucleótidos. La Figura 2.16. muestra un diagrama esquemático de rARN de oocitos de Xenopus laevis. También en este caso la el conocimiento de la estructura permitirá aclarar la función de cada una de las subunidades ribosomales y permitirá comprender las relaciones entre proteínas y ácidos nucleicos.
Figura 2.16. Diagrama esquemático del rARN de oocitos de Xenopus laevis.
a) Seudonudo de una región altamente conservada en RNA 16s. Las lineas más finas indican regiones escenciales para la función ribosomal
b) Región 23s ARN, de reconocimiento de la proteína L1. Las líneas finas indican tres pares de bases (que ocurren en la estructura terciaria). Esta información proviene de la información filogenética y de las reactividades químicas de las bases.
c) Pareamientos alternos en 28s-ARN de Xenopus laevis. La estructura de la izquierda
i) es consistente con las reactividades químicas del ARN libre mientras que la estructura de la derecha se apoya en datos a partir de subunidades intactas.Extraído de Gluick, T.C. and Draper, D.E. (1992) Current Opinion in Structural Biology 2: 338-344.
Para estas moléculas se ha descrito también otras funciones como una función autocatalítica observada por primera vez en el protozoo Tetrahymena.
2.2.2. Proteínas
El producto final del mensaje genético es una proteína. Las proteínas son macromoléculas lineales, formadas por subunidades ámino acídicas enlazadas cabeza -cola para formar polipéptidos de alto peso molecular. (Para un estudio detallado sobre estructura de proteínas revisar : H.Cid, Monografía " Estructura de Proteínas" (1995)). Las unidades monoméricas son los L-ámino ácidos. La estructura de Alanina se muestra en la figura 2.17 para ejemplificar la estereoquímica de los ámino ácidos que se encuentran en las proteínas. Su estructura química se describe en la Tabla 2.3.
Figura 2.17. Estereoquímica de D y L ámino ácidos. Arriba se representa la estructura en el sistema RS para la representación de la configuración absoluta y abajo la proyección de Fisher. Las figuras a la izquierda corresponden a R-Alanina o L(+) alanina y las de la derecha a S-Alanina o D(-)alanina.
Los L- ámino ácidos se unen a través de enlaces peptídicos, a través de una reacción de condensación cabeza-cola con pérdida de una molécula de agua, para formar un polipéptido.
Figura 2.18. Formación del enlace peptídico.
La existencia de un enlace peptídico, covalente doble parcial entre C y N, restringe la movilidad de la cadena polipeptídica principal, a sólo rotaciones alrededor del C y de los enlaces covalentes correspondientes a las cadenas laterales de cada uno de los ámino ácidos como se observa en la Figura 2.19:
Figura 2.19. Dimensiones del enlace peptídico. Distancias interatómicas incluyendo la longitud de los enlaces puente hidrógeno con un grupo peptídico vecino. Los átomos que están ubicados entre las lineas punteadas pertenecen a un plano.
Las características especiales de cada proteína están en su mayor parte codificadas en la secuencia de ámino ácidos, que es la señal transmitida por los genes. La secuencia de los ámino ácidos en la cadena polipeptídica de una proteína se denomina Estructura primaria. Por ejemplo, la estructura primaria de R-ficocianina de Synechococcus sp. se observa en la figura 2.20:
Figura 2.20. Secuencia ámino acídica de R-ficocianina de Synechococcus sp., una proteína accesoria del aparato fotosintético en algas uni y pluricelulares, de peso molecular 17335. Se utiliza el código de una letra.
Como ya se ha descrito, en todas las proteínas el enlace peptídico une a los ámino ácidos, conservando las características químicas de las cadenas laterales, es decir, su polaridad, tamaño y composición, y, en gran medida, cuando existen grupos ionizables, del estado de ionización de los ámino ácidos. Por ejemplo, si consideramos el ácido aspártico, su grupo carboxilo estará disociado a pH mayores que 4.0 (ver Tabla 2.3), y por lo tanto a ese pH tendrá mayores posibilidades de participar en una interacción electrostática.
Si no está disociado, sus posibilidades de formar uniones puente hidrógeno son mayores. Cuando la mitad de los carboxilos está disociada, se alcanza el equilibrio y este equilibrio está representado por los pK (-ln Keq) del grupo ionizable considerado.
Tabla 2.3.
Estructura química de los 20 amino ácidos presentes en las proteínas,
siglas más usadas y valores de pKa para sus grupos ionizables
Tabla 2.3.
Estructura química de los 20 amino ácidos presentes en las proteínas,
siglas más usadas y valores de pKa para sus grupos ionizables
Anexo 2.3
Punto isoeléctrico
Se denomina punto isoeléctrico al pH en el cual una proteína, péptido o ámino ácido (AA) no migran cuando están expuestos a un campo eléctrico. También puede definirse como el pH en que la forma predominante es AAo, en que cualquiera pequeña concentración de AA+ esté balanceada por una pequeña concentración de AA-.
Sean las dos reacciones correspondientes a la disociación de un ámino ácido.
Ka1 Ka2
AA+
AAo + H+ AAoAA- + H+
Ka1 = [AAo] [H+] Ka2 = [AA-] [H+]
[AA+] [AAo]
de estas ecuaciones despejamos
[AAo] = Ka1[AA+] y [H+] = Ka2[AAo]
[H+] [AA-]
[H+] = Ka1Ka2[AA+]
[H+][AA-]
[H+]2 = Ka2Ka1[AA+]
[AA-]
Si [AA+] = [AA-]
entonces
[H+] = (Ka1Ka2)1/2 o lo que es lo mismo
pH = pKa2 + pKa1
2
Es decir para un ámino ácido como alanina por ejemplo, el punto isoeléctrico corresponderá al promedio entre pKa1 y pKa2.
Este tratamiento puede ampliarse a los diferentes ámino ácidos y a las cadenas polipeptídicas considerando los grupos disociables y sus respectivos pK.
La figura representa el comportamiento de ionización de las cadenas laterales de ácido aspártico (D), ácido glutámico (E), histidina (H) Lisina (K) y arginina (R).
Figura 2.21. Comportamiento de ionización de las cadenas laterales de ámino ácidos.
El comportamiento de ionización tanto de ámino ácidos libres como en cadenas polipeptídicas puede determinarse mediante las curvas de titulación de los grupos cargados. Ejemplos de este tipo de curvas se muestran en las figuras 2.22 para alanina y 2.23 para ácido glutámico, histidina y lisina:
Figura 2.22. Titulación de alanina. Se indican las formas ionizadas en cada inflexión.
Figura 2.23. Curvas de titulación para ácido glutámico (...), histidina (__) y lisina (---).
Este comportamiento de ionización, hace que la estabilidad de las moléculas se vea afectada. Es decir, cambiando el pH del medio, la molécula considerada se acomodará a un nuevo mínimo de energía potencial que involucrará este estado de ionización (que a su vez afectará los factores electrostáticos, posibilidades de formación de puentes hidrógeno, solubilidad, etc).
Las características de cada ámino ácido, tamaño, polaridad, grado de hidrofobicidad, y la secuencia ámino acídica son elementos determinantes en la estructura tridimensional que adquiera la proteína.
A modo de ejemplo, se ha podido relacionar la presencia de ámino ácidos específicos asociados a rasgos estructurales y funcionales particulares:
Glicina
Proporciona un mínimo de impedimento estérico a la rotación y por lo tanto suele encontrarse en las vueltas en cadenas polipeptídicas.
Alanina, Valina, Leucina e Isoleucina
Participan en las interacciones hidrofóbicas que estabilizan una proteína.
Prolina
Este iminoácido contiene una amina secundaria lo que limita su flexibilidad conformacional. Tiene influencia en el plegamiento de la cadena polipeptídica.
Fenilalanina, Tirosina y Triptofano
Participan en núcleos hidrofóbicos y son capaces de estabilizar moléculas planas. Tirosina además posee un grupo funcional -OH; se le encuentra frecuentemente participando en la catálisis.
Serina y Treonina
Poseen un grupo funcional -OH, con propiedades ácidas débiles. Pueden formar ésteres con ácidos fosfóricos, es decir pueden constituir sitios de fosforilación de proteínas, y además por sus características pueden también constituir sitios de unión de azúcares en glicoproteínas (glicosilación)
Acidos Glutámico y Aspártico
Se encuentran disociados a pH neutro y proporcionan en general grupos aniónicos en la superficie de la proteína.
Asparragina y Glutamina
Son ámino ácidos polares con capacidad de interaccionar vía puentes hidrógeno.
Cisteína
Posee un grupo funcional -S-H, un ácido débil que se oxida espontáneamente en presencia de oxígeno y forma los denominados puentes disulfuro o -S-S-. Generalmente están presentes en proteínas solubles, y en gran número en aquellas sometidas a stress mecánico. Este tipo de enlace es muy importante en algunas proteínas ya que produce la estabilización de la estructura.
Metionina
Este ámino ácido se encuentra en baja cantidad en las proteínas y preferentemente se localiza en el interior de la proteína por su carácter hidrofóbico.
Histidina
Posee un grupo funcional básico capaz de aceptar un protón para formar el ácido conjugado. Se le ha encontrado formando parte del sitio activo de varias enzimas(tripsina) y en sitios de unión de metales(hemoglobina).
Lisina y Arginina
Presentan un grupo potencialmente reactivos y a pH neutro se encuentra casi siempre protonados. Arginina puede ser sitio de unión de fosfatos.
Si pudiésemos observar la cadena creciente en sus interacciones con el medio ambiente, veríamos una continua acomodación de la cadena polipeptídica, en una búsqueda de lograr un mínimo de energía. Esto implica lograr un plegamiento de la cadena polipeptídica que asegure una maximización de las interacciones para lograr una máxima estabilidad estructural y un reordenamiento del solvente para que produzca un máximo de entropía.
Estas interacciones producen finalmente la estructura de una proteína. Un modelo de la estructura de una proteína se observa en la Figura 2.24:
Figura 2.24. Modelo tridimensional de la estructura de una proteína globular. Se muestran sólo las posiciones de los C.
La Figura 2.24 muestra la dirección del plegamiento de una cadena polipeptídica y sólo se indican los Ca para simplificar la figura. La complejidad estructural de la macromoléculas, proteínas, ácidos nucleicos y polisacáridos ha dificultado el conocimiento de una información estructural detallada que permitiese entender la función de los sistemas. Los métodos en uso para el estudio estructural de las macromoléculas: Difracción de Rayos X, Resonancia Magnética Nuclear, Espectrometría de masas, polarización de fluorescencia, métodos de marcaje isotópico, y otros han debido utilizarse en conjunto para lograr obtener la información necesaria.
Ideas Importantes
- Las macromoléculas biológicas, o las moléculas grandes están formadas por subunidades monoméricas o bloques constitutivos.
- Todas ellas poseen tanto regiones polares como apolares. Tanto proteínas como ácidos nucleicos poseen grupos cargados, y su estabilidad es altamente dependiente de las condiciones del medio (pH, temperatura, presencia de sales, etc)
- En las macromoléculas lineales es posible identificar una secuencia o estructura primaria. De la secuencia y de los factores ambientales depende la estructura que adoptará la macromolécula en solución.
Cuestionario
1. ¿Qué propiedades fundamentales tienen los polisacáridos ramificados?
2. Mencione algunas funciones en las que participan los polisacáridos y discuta cómo se relaciona su estructura con la función.
3. ¿Qué grupos funcionales se encuentran en las cadenas laterales de los ámino ácidos? ¿Qué importancia tienen para la estructura de las proteínas
a) los grupos hidrofóbicos?
b) ¿los grupos polares?
c) ¿los grupos cargados?
4. Para el péptido L-Ala-L-His-L-Asp-L-Arg:
a) determine el punto isoeléctrico,
b) dibuje las formas ionizadas que existen a pH 5 y 9.
5. Para el siguiente péptido: (glicil-L-triptofanil-L-prolil-L-seril-L-lisina)
a) Escriba la estructura
b) A pH 7, este péptido migrará ¿hacia el cátodo o el ánodo?
6. Investigue las propiedades estructurales de la seda, la keratina y el colágeno.
7. ¿En qué se sustenta la estabilidad de los ácidos nucleicos?
Bibliografía Básica
1. Cid, H. Estructura de Proteínas. (1995) Proyecto de desarrollo de la Docencia. Universidad de Concepción.
2. Watson, J.D. Biología Molecular del Gen (1978) Fondo Educativo Interamerican, 3a edición.
3. Morrison, R.T and Boyd, R.N. Organic Chemistry.(1978) Allyn and Bacon Inc. 3rd edition.
4. Metzler, D. Biochemistry. (1977)Academic Press, New York
Bibliografía Avanzada
1. Cantor, C.R.and Schimmel, P.R. Biophysical Chemistry Parts 1, 2, 3.(1979), W.H.Freeman and Co.
2. Saenger, W. Principles of DNA Structure. Springer Verlag(1989)
3. Watson, D., and Crick, F.H. (1953), A structure of Deoxyribose nucleic acid. Nature 171: 737-738.
4. Crick, F.H. and Watson, J.D. (1954) The complementary structure of deoxyribonucleic acid. Proc. Roy. Soc (London) Ser A 223: 80-86